Forskjell mellom versjoner av «BioHackerLab»
(→Bioinformatikk) |
m (lagt til Kategorien Rom) |
||
| (168 mellomliggende revisjoner av 5 brukere er ikke vist) | |||
| Linje 1: | Linje 1: | ||
| − | + | Biologi gruppen på Bitraf heter Bioraf. | |
| + | |||
| + | See also our chat channel #biohackers on bitraf.slack.com. To join, send yourself an invitation at https://bitraf.no/slack-invite/ | ||
= Planlegging = | = Planlegging = | ||
| Linje 17: | Linje 19: | ||
* 13. juni 2016: http://www.meetup.com/bitraf/events/231692626/ - > Vi fikk besøk av Marius Øgaard fra Oslo Lifetech | * 13. juni 2016: http://www.meetup.com/bitraf/events/231692626/ - > Vi fikk besøk av Marius Øgaard fra Oslo Lifetech | ||
| + | |||
| + | * 27. juni 2016: Bruk av DNA-elektroforeseutstyret ble demonstrert, og de fleste fikk prøve pipettering av DNA til gel'en. Heikki presenterte utkast til brev til mulige sponsorer og tok i mot tilbakemeldinger. | ||
| + | |||
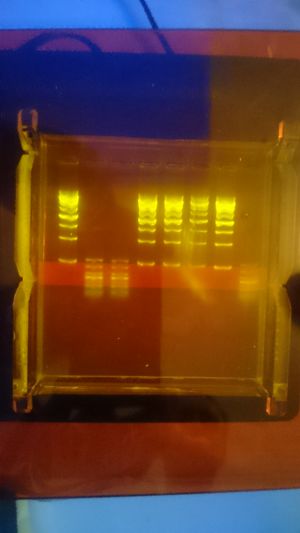
| + | [[Fil:Dsc 0069.jpg|miniatyr|sentrer|Resultat av gel-elektroforese demonstrert på meetup 27.06.16. Spor #1 og 4-7 fra venstre: Dongsheng Biotech 1kb ladder (~5 uL). Spor 2,3 og 8: Dongsheng Biotech 50bp ladder (~5 uL). Bildet er tatt av Heikki Sørum.]] | ||
| + | |||
| + | |||
| + | *11. juli 2016: http://www.meetup.com/bitraf/events/232455024/. We performed PCR using 3 different yeast samples. As it became pretty late, the PCR samples were put in the freezer for later electrophoresis and checking of the results. To have something to look at, we performed electrophoresis of samples from the previous two PCR runs. Update: The PCR samples were checked by electrophoresis July 24. 1 of 6 reactions was succesful. See https://bitraf.no/wiki/BioHackerLab/Experimental#11_Jul_2016_-_Bitraf_PCR_.233 | ||
| + | |||
| + | *Monday <strike>August 1</strike> August 8 2016: DIY Biolab: Using basic bioinformatics Tools; PyMOL: http://www.meetup.com/bitraf/events/232677504/ | ||
| + | |||
| + | *Monday August 22: DIY Biolab: A visit from the UiO iGEM team + strawberry DNA extraction: https://www.meetup.com/bitraf/events/233059010/ | ||
| + | |||
| + | *Monday September 5 2016: Bioraf byggekveld: http://www.meetup.com/bitraf/events/233816161/ We placed a book shelf in the main working space and put the equipment on it for easier access. | ||
| + | |||
| + | *Monday September 19 2016: Sequence Alignment Workshop: https://www.meetup.com/bitraf/events/234064840/ | ||
| + | |||
| + | =Planlagte meetups= | ||
| + | |||
| + | |||
= Videre jobbing = | = Videre jobbing = | ||
De av deltakerne som ønsket å jobbe med dette videre delte seg i to grupper som skal jobbe med Mikrobiologi og Utstyr til labben. Mikrobiologi-gruppen har planer for å jobbe med ølbrygging og klassifisering av gjær med [http://openpcr.org/ Open PCR]. | De av deltakerne som ønsket å jobbe med dette videre delte seg i to grupper som skal jobbe med Mikrobiologi og Utstyr til labben. Mikrobiologi-gruppen har planer for å jobbe med ølbrygging og klassifisering av gjær med [http://openpcr.org/ Open PCR]. | ||
| + | |||
| + | [[Fil:YeastPCR ITS ITS4 050716.jpg|miniatyr|sentrer|Result from PCR experiment 05 july 2016 to copy the 5.8S rRNA gene RDN58 and flanking ITS regions from yeast (S. cerevisae). Primers used were ITS1 (TCCGTAGGTGAACCTGCGG) and ITS4 (TCCTCCGCTTATTGATATGC). Primers were supplied by Macrogen Inc. Primer target concentration: 0.5 uM each. From left: DSBio 1kb ladder (5 uL), DSBio 50bp ladder (5 uL), PCR sample 1 (10 uL), PCR sample 2 (10 uL), PCR negative control (no template) sample (~5-10 uL). Electrophoresis at 75V for ~45 min on 1 % agarose with GelGreen DNA stain. Visualized with DarkReader DR22 transilluminator. PCR performed 05.07.16 with OpenPCR and DongSheng Biotech Taq mix. Reaction volume 50 uL. Template source is store bought dry yeast (Idun tørrgjær). Template source was prepared by dissolving 0.1 g dry yeast in 10 mL distilled water, and incubating 50 uL of the resulting yeast solution in a PCR tube at 98C for 10 min in openPCR. For PCR sample 1 and 2, 1 uL and 2 uL of the supernatant after incubation was added to the reaction mix, respectively. PCR program was as follows: Initital denaturation: 94C for 3 min. Repeated cycles: Denaturation: 94C for 30s. Anneal: 55.5C for 30s. Extension: 72C for 1 min. Final extension: 72C for 10 min. 35 cycles. Total run time: ~2h 20 min. ]] | ||
| + | |||
| + | ==Utstyr vi har== | ||
| + | * 1x OpenPCR | ||
| + | * Automatpipetter for områdene 1-10, 10-100 og 100-1000 uL. (2 sett) | ||
| + | *1x lavhastighets mikrosentrifuge for 1.5 mL-rør | ||
| + | *1x Vortex mixer | ||
| + | *Elektroforese-kammer: 2x Carolina deluxe electrophoresis chamber | ||
| + | *Elektroforesestrømforsyning: 1x BioRad PowerPac Basic. | ||
| + | *1x DarkReader DR22A blue light transilluminator | ||
| + | *Mikrobølgeovn | ||
| + | *Vanndestillasjonsapparat | ||
| + | *Vannbad | ||
| + | *Varmeplate med magnetrører | ||
| + | *Eppendorf Biophotometer 6131 | ||
| + | *Diverse pelistaltiske pumper | ||
| + | *Biohackacademy pelistaltisk pumpe | ||
| + | *NIKON Diaphot TMD mikroskop | ||
| + | |||
| + | [[Fil:OpenPCR.jpg|miniatyr|OpenPCR]] | ||
| + | |||
| + | [[Fil:Electrophoresis.jpg|miniatyr|Electrophoresis equipment: Carolina deluxe gel chamber and BioRad PowerPac power supply. Bottles with 10x TAE buffer and electrophoresis-grade agarose (small bottle) are also visible.]] | ||
| + | |||
| + | [[Fil:Pipettes.jpg|miniatyr|Set of micropipettes; 1-10, 10-100 and 100-1000 uL. LHP brand (Liquid Handling Products).]] | ||
| + | |||
| + | [[Fil:I016 Biophotometer.jpg|miniatyr|Eppendorf Biophotometer 6131. Used for quantification of nucleic acids.]] | ||
== Utstyr vi ønsker oss == | == Utstyr vi ønsker oss == | ||
| − | * | + | *pH-meter |
| − | * UV-kamera: | + | * UV-kamera. (Hva brukes dette til? --[[Bruker:Jarlemag|Jarlemag]] ([[Brukerdiskusjon:Jarlemag|diskusjon]]) 22. jul. 2016 kl. 18:41 (UTC)) |
| − | * | + | * Sentrifuge: Høyhastighetssentrifuge for 10 mL- og 50 mL-rør. Ex. Sorvall Biofuge Primo / Primo R. |
| − | |||
| − | |||
| − | |||
| − | |||
* Fryser (ideelt -20 C) | * Fryser (ideelt -20 C) | ||
| − | *Kjøleskap | + | * Kjøleskap |
| − | + | * Varmeskap med shaker. Eks: https://webshop.no.alere.com/222ds-benchtop-shaking-incubator-230v_1.aspx | |
| − | * Varmeskap med shaker | + | *Varmeblokk |
| − | * | + | * Autoklav |
| − | * | ||
* Lab-glass (flasker, rør m.m.) | * Lab-glass (flasker, rør m.m.) | ||
| + | *Målepipetter i glass | ||
| + | *Flere automatpipetter? | ||
* Filter-utstyr | * Filter-utstyr | ||
* Vekst-medier | * Vekst-medier | ||
| − | * Qubit | + | * Qubit Fluorometer |
| + | * Mikroskop med minst 800x forstørrelse (x400 luft, x800 oljeimmersjon), Confokalt og fase kontrast. https://no.wikipedia.org/wiki/Fasekontrastmikroskop f.eks. [https://www.dinkikkert.no/mikroskop/optika-b-500tiph] Optika B-500TiPh fra japanphoto | ||
| + | *Presisjonsvekt (F.eks Kern EMB 100-3: https://www.vektekspert.no/presisjonsvekt-kern-emb.html) | ||
| + | *Mikroplateleser, ex. Tecan Infinite 200 | ||
| − | I tillegg trenger vi | + | I tillegg trenger vi laboratoriemøbler/inventar som f.eks.: |
| + | * Stålbenk/utslagsvask | ||
| + | * Fumehood / labbenk | ||
| + | * Filter til Fumehood | ||
| + | *Oppvaskmaskin | ||
| + | |||
| + | ==Mikroskopi== | ||
| + | |||
| + | ''Content: Roland van Dierendonck og Arne Andersen (Limnoan) '' | ||
| + | |||
| + | At Bitraf we have the NIKON Diaphot TMD inverted microscope. | ||
| + | Always cover the microscope after using to avoid dust on the lenses. | ||
| + | A manual of the NIKON Diaphot TMD can be found here: https://neurophysics.ucsd.edu/Manuals/Nikon/Diaphot-TMD.pdf | ||
| + | More links on this webpage from Arizona edu: https://lavinia.as.arizona.edu/~mtuell/scopes/Diaphot.php - according to the author, this instrument was used to create the first test-tube baby! | ||
| + | |||
| + | [[Fil:Mikroskop.png|miniatyr|sentrer|Mikroskop deler fra handleding ]] | ||
| + | |||
| + | '''Fasekontrastmikroskopi''': Fasekontrast er en metode for å gjøre mikroskop-preparat bedre synlige uten å farge dem. | ||
| + | Rent praktisk brukes sett av matchende ringfilter. | ||
| + | Det ene sitter på kondensatoren, det andre inne i objektivet. | ||
| + | Filteret på kondensatoren lager en ring av lys. Størrelsen på ringen skal passe med en tilsvarende ring i objektivet. Når alt er riktig innstilt, skal de to ringene overlappe hverandre. | ||
| + | '''Denne innstillingen skal ikke endres av brukeren!''' | ||
| + | Som nevnt, kommer filterne i par: På kondensatoren er de merket med PhL – Ph3. Høyere tall står for høyere forstørrelse på objektivet. Objektivene er merket på tilsvarende måte, men det er vanskelig å se på vårt mikroskop. | ||
| + | Vanligvis bytter en filter og objektiv samtidig, men mismatch kan av og til gi interessante effekter. | ||
| + | https://en.wikipedia.org/wiki/Phase-contrast_microscopy | ||
| + | |||
| + | '''Phase-contrast microscopy''': Phase-contrast is a method of making microscope specimens more visible without staining them. | ||
| + | In practical terms, sets of matching ring filters are used. One is on the condenser, the other inside the lens. The filter on the condenser creates a ring of light. The size of the ring should fit with a corresponding ring in the lens. | ||
| + | When everything is set correctly, the two rings should overlap. | ||
| + | '''This setting should not be changed by the user!''' | ||
| + | As mentioned, the filters come in pairs: On the condenser they are marked with PhL - Ph3. Higher numbers represent higher magnification of the lens. The lenses are marked in a similar way, but it is difficult to see on our microscope. Usually, a filter and lens change at the same time, but a mismatch can sometimes give interesting effects. | ||
| + | https://en.wikipedia.org/wiki/Phase-contrast_microscopy | ||
| + | |||
| + | '''Mørkfelt''':Mikroskopet på Bitraf har antakelig ikke denne innstillingen. | ||
| + | Mørkfelt betyr at en lager en skygge, som dekker hele synsfeltet. På et vanlig mikroskop kan en for eksempel legge en mynt på lampa. Forskningsmikroskop har ofte mørkfelt som et valg sammen med fasekontrast. | ||
| + | Når en legger et objekt på mikroskopet, vil noe av lyset brytes, slik at objektene blir lyse på mørk bakgrunn. | ||
| + | https://en.wikipedia.org/wiki/Dark-field_microscopy | ||
| + | |||
| + | '''Dark field''': The microscope on Bitraf probably does not have this setting. | ||
| + | Dark field means that you create a shadow, which covers the entire field of view. On an ordinary microscope, for example, you can put a coin on the lamp. Research microscopes often have dark fields as a choice along with phase contrast. | ||
| + | When you place an object on the microscope, some of the light will be refracted, so that the objects become light on a dark background. | ||
| + | https://en.wikipedia.org/wiki/Dark-field_microscopy | ||
| + | |||
| + | |||
| + | '''Mikroskopkurs''' | ||
| + | |||
| + | ''Planlagt av Roland van Dierendonck og Arne Andersen (Limnoan).'' | ||
| + | |||
| + | En mikroskopkurs varer 2 eller 3 timer og er notvendig for å bruke mikroskop. I kursen vi lære mikroskopdeler, og også å lage mikroskoppreparater. | ||
| + | |||
| + | Kurs inhold: | ||
| + | |||
| + | Hva du IKKE må røre (what NOT to touch! This is important because the microscope is a very expensive and delicate instrument and some parts are better left unchanged): | ||
| + | * Condensator screws | ||
| + | * Condensator focus knob (knotten med ord "TENSION") | ||
| + | * Lenses (don't touch the lenses with your fingers / ikke rør linser) | ||
| + | * Keep the dust cover on when not in use / la støvhette på | ||
| + | |||
| + | Parts to use / Deler vi bruker: | ||
| + | * X&Y-axis knobs / XY-bord bevegelse knot | ||
| + | * diaphragm / blenner | ||
| + | * Phase-contrast condenser / fase-kontrast kondensator | ||
| + | * Fokus knobs (coarse and fine) / fokus knot (grov og fint) | ||
| + | * Binocular eye piece distance / okular | ||
| + | * "Optical path change-over knob" for directing light to ocular or camera | ||
| + | * Front camera port / kameraport | ||
| + | |||
| + | Maintenance / Vedlikehold: | ||
| + | * Both the lenses and the phase-contrast filters are very delicate. Clean by blowing air with a rubber air blower. | ||
| + | |||
| + | Theory Resources (English og Norsk) | ||
| + | * Mikroskopi Presentasjon fra BioHack Academy (PDF): http://biohackacademy.github.io/bha6/class/4/pdf/4.1%20Microscopy%20and%20Optics.pdf | ||
| + | * Enkel Mikroskopi av Arne Andersen (Limnoan) i ''Biolog'' (PDF): https://arena-attachments.s3.amazonaws.com/12048616/1b6de336971678994fa83c4be560562e.pdf?1621853707 | ||
| + | * En enkel metode for planteplanktonundersøkelse av Arne Andersen (Limnoan): https://arena-attachments.s3.amazonaws.com/12048615/5c491b7bcc512441bb9d35fbaa7e32dd.pdf?1621853700 | ||
| + | |||
| + | This is a Nikon microscope and a Nikon camera body can be attached to make photos and films. | ||
| + | For other cameras adapters can be designed and 3D printed. | ||
| + | |||
| + | Mikroskoputstyr vi ønsker oss: | ||
| + | * Bedre støvhette | ||
| + | * Rubber air blower | ||
| + | |||
| + | Things to arrange: | ||
| + | * When is the course going to be (date and time) | ||
| + | * Max people that can join (2/3) | ||
| + | * Can we issue a Badge in the P2k16 system of Bitraf so partipants get one when they did the course? | ||
==PCR prosjekt== | ==PCR prosjekt== | ||
| Linje 52: | Linje 185: | ||
*Sammensetning og lengde av DNA-tråden mellom start- og sluttpunktet kan variere. Informajson om lengden av DNA-fragmentene (visualiseres ved gel-elektroforese), og om kopiering fant sted (positiv/negativ reaksjon) kan brukes til å gjøre enkle genetiske analyser. | *Sammensetning og lengde av DNA-tråden mellom start- og sluttpunktet kan variere. Informajson om lengden av DNA-fragmentene (visualiseres ved gel-elektroforese), og om kopiering fant sted (positiv/negativ reaksjon) kan brukes til å gjøre enkle genetiske analyser. | ||
*Opparbeidet DNA kan (gitt god nok mengde og kvalitet) sendes til nærmere analyse av DNA-sekvensen (sekvensering). | *Opparbeidet DNA kan (gitt god nok mengde og kvalitet) sendes til nærmere analyse av DNA-sekvensen (sekvensering). | ||
| + | |||
| + | PCR visualization: https://www.youtube.com/watch?v=2KoLnIwoZKU | ||
===Hvorfor gjøre PCR?=== | ===Hvorfor gjøre PCR?=== | ||
| Linje 58: | Linje 193: | ||
*En klassisk molekylærbiologi-teknikk. I daglig bruk verden over. Uunnværlig for molekylærbiologisk forskning og medisinsk diagnostikk. Nobelpris-vinnende. | *En klassisk molekylærbiologi-teknikk. I daglig bruk verden over. Uunnværlig for molekylærbiologisk forskning og medisinsk diagnostikk. Nobelpris-vinnende. | ||
*Potensiale for praktiske anvendelser med allmenn interesse. F.eks kontroll av artsopprinnelse for matvarer. | *Potensiale for praktiske anvendelser med allmenn interesse. F.eks kontroll av artsopprinnelse for matvarer. | ||
| − | |||
===Hva trengs for PCR?=== | ===Hva trengs for PCR?=== | ||
| Linje 176: | Linje 310: | ||
*http://no.frederiksen.eu/inspirasjon/biologi/bioteknologi/tips-og-raad | *http://no.frederiksen.eu/inspirasjon/biologi/bioteknologi/tips-og-raad | ||
*http://www.naturfag.no/utstyrsbeskrivelse/vis.html?tid=709639 | *http://www.naturfag.no/utstyrsbeskrivelse/vis.html?tid=709639 | ||
| − | + | *https://seqcore.brcf.med.umich.edu/sites/default/files/html/pcr.html | |
DNA Learning Center Biology Animation Library - Polymerase Chain Reaction: https://www.dnalc.org/resources/animations/pcr.html | DNA Learning Center Biology Animation Library - Polymerase Chain Reaction: https://www.dnalc.org/resources/animations/pcr.html | ||
| Linje 193: | Linje 327: | ||
https://www.kickstarter.com/projects/563115656/3d-printer-into-pcr-machine-conversion | https://www.kickstarter.com/projects/563115656/3d-printer-into-pcr-machine-conversion | ||
| − | Arduino PCR thermal cycler for under $85: http://www.instructables.com/id/Arduino-PCR-thermal-cycler-for-under-85/?ALLSTEPS | + | [[Arduino]] PCR thermal cycler for under $85: http://www.instructables.com/id/Arduino-PCR-thermal-cycler-for-under-85/?ALLSTEPS |
"Coffee Cup PCR": http://www.instructables.com/id/Coffee-Cup-PCR-Thermocycler-costing-under-350/?ALLSTEPS | "Coffee Cup PCR": http://www.instructables.com/id/Coffee-Cup-PCR-Thermocycler-costing-under-350/?ALLSTEPS | ||
| Linje 216: | Linje 350: | ||
MEATF + MEATR [2] | MEATF + MEATR [2] | ||
| − | HorseSSR-FWD + HorseSSR-REV [2] | + | HorseSSR-FWD + HorseSSR-REV [2]: |
| + | |||
| + | FWD: 5'-TTC TGC TCT GGG TGT GCT ACT T-3' (22mer) | ||
| + | REV: 5'-CTA CTT CAG CCA GAT CAG GC-3' (20mer) | ||
S-D-Bact-0515-a-A-19 + S-D-Bact-0341-b-S-17 [1] | S-D-Bact-0515-a-A-19 + S-D-Bact-0341-b-S-17 [1] | ||
| + | |||
| + | S-D-Bact-0515-a-A-19: 5'-TTA CCG CGG CTG CTG GCA C-3' (19mer) | ||
| + | S-D-Bact-0341-b-S-17: 5'-CCT ACG GGN GGC WGC AG-3' (17mer) | ||
| + | |||
| + | |||
[1]: See http://openwetware.org/wiki/User:Jarle_Pahr/16S_RNA | [1]: See http://openwetware.org/wiki/User:Jarle_Pahr/16S_RNA | ||
| Linje 231: | Linje 373: | ||
ITS 1 (5' TCCGTAGGTGAACCTGCGG 3') + ITS4 (5' TCCTCCGCTTATTGATATGC 3'). " In the present study, the restriction patterns generated from the region spanning the internal transcribed spacers (ITS1 and ITS2) and the 5.8S rRNA gene were used to identify a total of 132 yeast species belonging to 25 different genera, including teleomorphic and anamorphic ascomycetous and basidiomycetous yeasts." (http://www.ncbi.nlm.nih.gov/pubmed/10028278, full text available through ResearchGate). | ITS 1 (5' TCCGTAGGTGAACCTGCGG 3') + ITS4 (5' TCCTCCGCTTATTGATATGC 3'). " In the present study, the restriction patterns generated from the region spanning the internal transcribed spacers (ITS1 and ITS2) and the 5.8S rRNA gene were used to identify a total of 132 yeast species belonging to 25 different genera, including teleomorphic and anamorphic ascomycetous and basidiomycetous yeasts." (http://www.ncbi.nlm.nih.gov/pubmed/10028278, full text available through ResearchGate). | ||
| + | |||
| + | ITS1 and ITS4 available (10 USD for 100 reactions) from The ODIN: http://www.the-odin.com/fungal-its-pcr-primers-for-identification-and-barcoding/ | ||
| Linje 250: | Linje 394: | ||
For PCR from pJP-1, replace pSB-seqA with pJP-1_seq5 (binding site is upstream of AgeI, as such this site is preserved from pSB-mg1, and this primer combination can also be used with pSB-mg1 for a longer PCR fragment) | For PCR from pJP-1, replace pSB-seqA with pJP-1_seq5 (binding site is upstream of AgeI, as such this site is preserved from pSB-mg1, and this primer combination can also be used with pSB-mg1 for a longer PCR fragment) | ||
| + | =Helse, miljø og sikkerheit (HMS)= | ||
| + | |||
| + | http://www.ncbi.nlm.nih.gov/books/NBK55884/ | ||
| − | = | + | ==El-sikkerhet== |
| + | https://www.physics.ohio-state.edu/~p616/safety/fatal_current.html | ||
==Ved nødsfall== | ==Ved nødsfall== | ||
| Linje 378: | Linje 526: | ||
===DMSO=== | ===DMSO=== | ||
| + | |||
| + | ===Guanidinium chloride=== | ||
| + | |||
| + | Finnes i mange kommersielle kit for DNA-rensing. Typisk konsentrasjon kan være 5 M: http://openwetware.org/wiki/Qiagen_Buffers | ||
| + | |||
| + | http://echa.europa.eu/information-on-chemicals/cl-inventory-database/-/discli/details/52449 | ||
| + | |||
| + | Faresetninger (Konsentrasjonsgrenseverdier for klassifisering som farlig avfall iht. COMMISSION REGULATION (EU) No 1357/2014): | ||
| + | *H302: 25% | ||
| + | *H315: 20% | ||
| + | *H319: 20% | ||
| + | |||
| + | Minste grenseverdi: 20% | ||
| + | |||
| + | Basert på dette vil en 5M løsning sannsynligvis regnes som farlig avfall. | ||
| + | |||
| + | ===Isopropanol=== | ||
| + | |||
| + | https://echa.europa.eu/information-on-chemicals/cl-inventory-database/-/discli/details/22308 | ||
| + | |||
| + | Faresetninger (Konsentrasjonsgrenseverdier for klassifisering som farlig avfall iht. COMMISSION REGULATION (EU) No 1357/2014): | ||
| + | *H225: " If the presence of [the] substance indicates that the waste is flammable, it shall be classified as hazardous by HP 3" | ||
| + | *H319 (20%) | ||
| + | *H336 (no limit given) | ||
==Aktuelle stoffblandinger== | ==Aktuelle stoffblandinger== | ||
| Linje 389: | Linje 561: | ||
EDTA: <1%. | EDTA: <1%. | ||
| − | Jf. begrensende grenseverdier for Tris, Eddiksyre og EDTA basert på disses faresetninger og lik henholdsvis 20%, 1% og 10% vil innholdet av Tris og eddiksyre dermed sannsynligvis medføre at konsentrert, ufortynnet buffer må klassifiseres som farlig avfall ved avhending. | + | Jf. begrensende grenseverdier for Tris, Eddiksyre og EDTA basert på disses faresetninger og lik henholdsvis 20%, 1% og 10% vil innholdet av Tris og eddiksyre dermed sannsynligvis medføre at konsentrert, ufortynnet 50X TAE buffer må klassifiseres som farlig avfall ved avhending. Se Aktuelle kjemikalier. |
| − | Konsentrert buffer fortynnes typisk | + | For 10X TAE buffer vil de aktuelle konsentrasjonene være ca.: |
| + | |||
| + | Tris: ~5% | ||
| + | Eddiksyre: 1,2% | ||
| + | EDTA: <0.2% | ||
| + | |||
| + | På grunn av innholdet av eddiksyre vil dermed også 10X TAE buffer sannynligvis klassifiseres som farlig avfall ved avhending. | ||
| + | |||
| + | Konsentrert buffer fortynnes typisk til 1x før bruk. Brukt buffer vil dermed sannsynligvis ikke inneholde konsentrasjoner av de nevnte stoffene over grenseverdiene, og kan i såfall avhendes som vanlig avfall. (OBS: Det er ikke tillatt å fortynne farlig avfall med den hensikt å komme under grenseverdiene). | ||
'''Påkrevd piktogram iht. CLP: GHS07''' | '''Påkrevd piktogram iht. CLP: GHS07''' | ||
==Sikkerhetsdatablader== | ==Sikkerhetsdatablader== | ||
| + | |||
| + | EU-krav til sikkerhetsdatablader er gitt i Annex II til REACH: http://eur-lex.europa.eu/legal-content/en/TXT/PDF/?uri=CELEX:02006R1907-20160401 | ||
| + | |||
| + | Se også EHCA Guidance on the compilation of safety data sheets: http://echa.europa.eu/documents/10162/13643/sds_en.pdf | ||
GelGreen: http://biotium.com/wp-content/uploads/2013/07/MSDS-41005.pdf | GelGreen: http://biotium.com/wp-content/uploads/2013/07/MSDS-41005.pdf | ||
| Linje 417: | Linje 601: | ||
==Faremerking== | ==Faremerking== | ||
| + | |||
| + | https://www.pervaco.no/skilt-fundament/ghs-clp-skilt | ||
http://www.miljodirektoratet.no/no/Publikasjoner/Publikasjoner/2011/Mars/Klassifisering_og_merking_i_CLP/ | http://www.miljodirektoratet.no/no/Publikasjoner/Publikasjoner/2011/Mars/Klassifisering_og_merking_i_CLP/ | ||
| Linje 439: | Linje 625: | ||
=Lenker= | =Lenker= | ||
| + | |||
| + | ==Lab techniques== | ||
| + | |||
| + | Alkaline lysis: http://bitesizebio.com/180/the-basics-how-alkaline-lysis-works/ | ||
==Andre grupper og nettsteder== | ==Andre grupper og nettsteder== | ||
| + | |||
| + | http://www.socializedscience.com/projects1.html | ||
| + | |||
BioHack Academy: https://biohackacademy.github.io/ | BioHack Academy: https://biohackacademy.github.io/ | ||
| Linje 484: | Linje 677: | ||
http://www.reach-chemconsult.com/en/seiten/ghs-konverter.html | http://www.reach-chemconsult.com/en/seiten/ghs-konverter.html | ||
| + | |||
| + | http://www.the-scientist.com/?articles.view/articleNo/14477/title/An-Accident-Waiting-to-Happen-/ | ||
'''Elektroforese:''' | '''Elektroforese:''' | ||
| Linje 492: | Linje 687: | ||
http://ehs.unl.edu/sop/s-electrophoresis_safety.pdf | http://ehs.unl.edu/sop/s-electrophoresis_safety.pdf | ||
| + | |||
| + | https://www.admin.ox.ac.uk/safety/policy-statements/s11-07/ | ||
http://www.labmanager.com/lab-health-and-safety/2010/05/electrophoresis-safety-tips?fw1pk=2#.V2Wh3_mLRD8 | http://www.labmanager.com/lab-health-and-safety/2010/05/electrophoresis-safety-tips?fw1pk=2#.V2Wh3_mLRD8 | ||
| + | |||
| + | https://www.admin.ox.ac.uk/safety/policy-statements/s11-07/ | ||
| + | |||
| + | http://www.di.uq.edu.au/sparq/RAs/SPARQedDNARNAElectrophoresisRA.pdf | ||
==Lover og forskrifter== | ==Lover og forskrifter== | ||
| Linje 501: | Linje 702: | ||
===Norske forskrifter=== | ===Norske forskrifter=== | ||
| + | |||
| + | Forskrift om særavgifter (relevant mht. bruk av teknisk sprit): http://lovdata.no/forskrift/2001-12-11-1451/§3-3-10 | ||
| + | |||
Forskrift om utforming og innretning av arbeidsplasser og arbeidslokaler (arbeidsplassforskriften) - Kapittel 8. Arbeid i omgivelser som kan medføre eksponering for biologiske faktorer: http://lovdata.no/forskrift/2011-12-06-1356/§8-1 | Forskrift om utforming og innretning av arbeidsplasser og arbeidslokaler (arbeidsplassforskriften) - Kapittel 8. Arbeid i omgivelser som kan medføre eksponering for biologiske faktorer: http://lovdata.no/forskrift/2011-12-06-1356/§8-1 | ||
| Linje 574: | Linje 778: | ||
Exploring Personal Genomics: http://www.amazon.com/Exploring-Personal-Genomics-Joel-Dudley/dp/0199644497?ie=UTF8&psc=1&redirect=true&ref_=oh_aui_detailpage_o06_s00 | Exploring Personal Genomics: http://www.amazon.com/Exploring-Personal-Genomics-Joel-Dudley/dp/0199644497?ie=UTF8&psc=1&redirect=true&ref_=oh_aui_detailpage_o06_s00 | ||
| + | The Art of Fermentation: An In-Depth Exploration of Essential Concepts and Processes from around the World: http://www.amazon.com/Art-Fermentation-Depth-Exploration-Essential/dp/160358286X?ie=UTF8&psc=1&redirect=true&ref_=oh_aui_detailpage_o06_s00 | ||
| − | + | Budding Yeast: A Laboratory Manual: http://www.cshlpress.com/default.tpl?action=full&--eqskudatarq=1071 | |
===Artikler=== | ===Artikler=== | ||
| Linje 590: | Linje 795: | ||
'''DNA barcoding:''' | '''DNA barcoding:''' | ||
| + | |||
| + | http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0066213 | ||
Nuclear ribosomal internal transcribed spacer (ITS)region as a universal DNA barcode marker for | Nuclear ribosomal internal transcribed spacer (ITS)region as a universal DNA barcode marker for | ||
| Linje 604: | Linje 811: | ||
==Kommersielt tilgjengelig utstyr== | ==Kommersielt tilgjengelig utstyr== | ||
| − | |||
http://www.edvotek.com/Equipment | http://www.edvotek.com/Equipment | ||
http://no.frederiksen.eu/ | http://no.frederiksen.eu/ | ||
| + | |||
| + | Anmeldelser, artikler, etc.: http://www.selectscience.net/ | ||
===PCR=== | ===PCR=== | ||
| Linje 619: | Linje 827: | ||
DarkReader Blue Light Transilluminator: http://www.clarechemical.com/transilluminator.htm | DarkReader Blue Light Transilluminator: http://www.clarechemical.com/transilluminator.htm | ||
| + | |||
| + | ===Spektro-/foto-/fluorometri=== | ||
| + | |||
| + | Qubit 3.0 fluorometer:https://www.thermofisher.com/order/catalog/product/Q33216. Listepris 15 340,00 NOK. | ||
| + | |||
| + | Spektrofotometer V1100D: http://no.frederiksen.eu/nettbutikk/felles-laboratorieutstyr/maaleutstyr/spektrofotometer-v1100d. Listepris 5 995,00 kr eks. MVA. | ||
| + | |||
| + | http://www.ebay.com/itm/Visible-Spectrometer-Laboratory-Spectrophotometer-220V-350-1020nm-721-/401130883021?hash=item5d654387cd:g:ONoAAOSwMmBVoPei. Listepris ca. NOK 2,329.51. | ||
| + | |||
| + | ===Annet laboratorieutstyr=== | ||
| + | |||
| + | http://no.frederiksen.eu/shop/product/termostatblokk--labnet--digital--enkel | ||
| + | |||
| + | http://no.frederiksen.eu/shop/product/bakteriedyrkingsskap-cultura | ||
==Kommersielt tilgjengelige reagenser og forbruksvarer== | ==Kommersielt tilgjengelige reagenser og forbruksvarer== | ||
| + | |||
| + | ===Mikrobiologi=== | ||
| + | |||
| + | Peptone LP0037: http://www.oxoid.com/UK/blue/prod_detail/prod_detail.asp?pr=LP0037 | ||
| + | |||
| + | Oxoid yeast extract: http://www.oxoid.com/UK/blue/prod_detail/prod_detail.asp?pr=LP0021&c=UK&lang=EN | ||
| + | |||
===PCR=== | ===PCR=== | ||
| Linje 632: | Linje 861: | ||
http://pearlbiotech.com/ | http://pearlbiotech.com/ | ||
| − | == | + | ===Restriksjonsenzymer=== |
| + | |||
| + | EcoRI: | ||
| + | |||
| + | Gjenkjenningssekvens (plusstråd/komplementær): GAATTC / GAATTC | ||
| + | |||
| + | *http://no.frederiksen.eu/shop/product/restriksjonsenzym-ecori | ||
| + | *https://www.neb.com/products/r0101-ecori | ||
| + | |||
| + | CfoI: | ||
| + | |||
| + | Gjenkjenningssekvens (plusstråd/komplementær): GCGC / GCGC | ||
| + | |||
| + | *https://no.promega.com/products/cloning-and-dna-markers/restriction-enzymes/cfoi/ | ||
| + | |||
| + | HaeIII: | ||
| + | |||
| + | Gjenkjenningssekvens (plusstråd/komplementær): GGCC / GGCC | ||
| + | |||
| + | *https://www.neb.com/products/r0108-haeiii | ||
| − | Forbruksvarer og reagenser: | + | HinfI: |
| + | |||
| + | Gjenkjenningssekvens (plusstråd/komplementær): GANTC/GANTC | ||
| + | |||
| + | https://www.neb.com/products/r0155-hinfi | ||
| + | |||
| + | Relevant litteratur: | ||
| + | |||
| + | Clark et al. Extended stability of restriction enzymes at ambient temperatures. Biotechniques. 2000 Sep;29(3):536-8, 540, 542.: http://www.ncbi.nlm.nih.gov/pubmed/10997268 | ||
| + | |||
| + | ==Leverandører av utstyr, tjenester og forbruksvarer== | ||
| + | |||
| + | ===Forbruksvarer og reagenser=== | ||
| + | |||
| + | http://www.metabion.com/products/index.php | ||
http://www.the-odin.com/ | http://www.the-odin.com/ | ||
| Linje 642: | Linje 904: | ||
http://dongshengbio.com/en/index.asp | http://dongshengbio.com/en/index.asp | ||
| − | http://www.onlinesciencemall.com/ (Selger bl.a. TAE | + | http://www.onlinesciencemall.com/ (Selger bl.a. TAE buffer) |
| − | Oligomersyntese | + | http://www.abpbio.com/product/nucleic-acid-solution-quantitation-kits/ |
| + | |||
| + | https://zageno.com/ | ||
| + | |||
| + | https://www.theconsumablescompany.com/ | ||
| + | |||
| + | ===Oligomersyntese=== | ||
http://macrogen.com/eng/ | http://macrogen.com/eng/ | ||
| Linje 652: | Linje 920: | ||
http://www.thermofisher.com/no/en/home/products-and-services/product-types/primers-oligos-nucleotides/invitrogen-custom-dna-oligos.html | http://www.thermofisher.com/no/en/home/products-and-services/product-types/primers-oligos-nucleotides/invitrogen-custom-dna-oligos.html | ||
| − | Sekvensering: | + | ===Sekvensering=== |
| + | |||
| + | '''General advice:''' | ||
| + | |||
| + | http://www.bgi.com/services/genomics/sanger-sequencing/single-sample-sequencing/ | ||
| + | |||
| + | http://www.nucleics.com/DNA_sequencing_support/sequencing-service-choosing.html | ||
| − | https://www.gatc-biotech.com/en/products/sanger-services/lightrun-sequencing.html | + | http://www.nucleics.com/DNA_sequencing_support/sequencing-service-reviews.html |
| + | |||
| + | '''GATC:''' | ||
| + | |||
| + | LightRun sequencing: https://www.gatc-biotech.com/en/products/sanger-services/lightrun-sequencing.html' | ||
| + | |||
| + | *Prepaid labels | ||
| + | *Premixed DNA and primer | ||
| + | *4.00 EUR / reaction, minimum order of 100 | ||
| + | *(Welcome offer: 50 reactions for 3.50 EUR each) | ||
| + | |||
| + | SUPREMErun sequencing: https://www.gatc-biotech.com/en/products/sanger-services/supremerun-sequencing.html | ||
| + | |||
| + | '''Macrogen:''' | ||
http://macrogen.com/eng/ | http://macrogen.com/eng/ | ||
| + | |||
| + | http://dna.macrogen.com/eng/support/ces/guide/order_guide.jsp | ||
| + | |||
| + | Standard sequencing: https://dna.macrogen.com/eng/order/ces/std/s_new_step1.jsp | ||
| + | *5.99 EUR/reaction (express, 24 turnaround) | ||
| + | * 4.5 EUR/reaction (regular, 4-5 workdays turnaround) | ||
| + | *+2 EUR/reaction for purification | ||
| + | |||
| + | EZseq: http://dna.macrogen.com/eng/order/ces/ezseq/ezseq_step1.jsp | ||
| + | |||
| + | *EZseq single direct: 4.5 EUR/label (minimum order of 50). | ||
| + | *24hr turnaround | ||
| + | |||
| + | Sample purification is not offered with EZseq. | ||
| + | |||
| + | Eco-seq: http://dna.macrogen.com/eng/order/ces/ecoseq/ecoseq_step1.jsp | ||
| + | |||
| + | *Eco-seq single direct: 4.99 EUR/label (minimum order of 50) | ||
| + | *Eco-seq single purification: 6.99 EUR/label | ||
| + | |||
| + | Custom sequencing: https://dna.macrogen.com/eng/support/ces/customized_seq_intro.jsp | ||
| + | |||
| + | Sample preparation guide: https://dna.macrogen.com/eng/support/ces/guide/ces_sample_prep.jsp | ||
| + | |||
| + | From FAQ: "Minimum 20ul of 100ng/ul(plasmids, unpurified PCR products) or 50ng/ul(purified PCR products) are required for a couple of reactions." | ||
| + | |||
| + | Sample submission guide: https://dna.macrogen.com/eng/support/ces/guide/ces_sample_submission.jsp | ||
| + | |||
| + | '''BaseClear:''' | ||
| + | |||
| + | See http://www.baseclear.com/genomics/sanger-sequencing | ||
| + | |||
| + | See https://orders.baseclear.com/ | ||
| + | |||
| + | Single run sequencing services: | ||
| + | |||
| + | Prepaid barcode sequencing: http://www.baseclear.com/genomics/sanger-sequencing/prepaid-barcode-sequencing | ||
| + | *DNA purification and premixing with primer is done by the customer | ||
| + | |||
| + | Quickshot: http://www.baseclear.com/genomics/sanger-sequencing/quick-shot | ||
| + | |||
| + | Primers options: | ||
| + | *Separate sample and primer or premix possible | ||
| + | *Free usage of universal primers (see standard primer list) | ||
| + | *Custom primer, sent with the order (10 pmol/uL in volume >20 uL (enough for max.10 reactions) | ||
| + | |||
| + | Sample options: | ||
| + | *Bacteria for plasmid isolation (miniprep) on a agar plate or as glycerol stock | ||
| + | *Purified plasmid in a minimal volume of 30 ul with a DNA concentration of 50 – 200 ng/ul | ||
| + | *Purified or Raw PCR product in a minimal volume of 30 ul with a DNA concentration >5 ng/μl | ||
| + | |||
| + | Pricing: ?? | ||
| + | Payment: Invoice by e-mail. | ||
| + | |||
| + | |||
| + | '''Source BioScience:''' | ||
| + | |||
| + | |||
| + | http://www.lifesciences.sourcebioscience.com/genomic-services/sanger-sequencing-service/ | ||
| + | |||
| + | http://www.lifesciences.sourcebioscience.com/genomic-services/sanger-sequencing-service/information/sample-requirements/ | ||
| + | |||
| + | 9.5 EUR/reaction for minimum order of 50 (475 EUR) | ||
| + | |||
| + | Concentration requirement for (purified) PCR product: 1ng/µl per 100bp | ||
| + | |||
| + | *Payment by credit card available. | ||
==DIY/Open hardware== | ==DIY/Open hardware== | ||
| Linje 664: | Linje 1 018: | ||
https://www.bento.bio/ | https://www.bento.bio/ | ||
| + | https://www.chaibio.com/ | ||
| + | |||
| + | http://hackteria.org/wiki/index.php/DIY_NanoDrop | ||
| + | |||
| + | http://www.thingiverse.com/thing:73910 | ||
| + | |||
| + | http://www.gaudi.ch/OpenDrop/ | ||
| + | |||
| + | http://www.instructables.com/id/DIY-BioPrinter/ | ||
| + | |||
| + | http://opentrons.com/ | ||
| + | |||
| + | https://github.com/biohackacademy | ||
==Prosjekter til inspirasjon== | ==Prosjekter til inspirasjon== | ||
| Linje 669: | Linje 1 036: | ||
http://www.instructables.com/id/DIY-Bio-plastics/ | http://www.instructables.com/id/DIY-Bio-plastics/ | ||
| + | ==Diverse== | ||
| + | |||
| + | http://dna-view.com/ | ||
=Bioinformatikk= | =Bioinformatikk= | ||
| + | |||
| + | ==Genomikk== | ||
| + | |||
| + | http://www.ncbi.nlm.nih.gov/projects/genome/assembly/grc/info/definitions.shtml | ||
==Databaser== | ==Databaser== | ||
[http://www.ncbi.nlm.nih.gov/genbank/ GenBank] | [http://www.ncbi.nlm.nih.gov/genbank/ GenBank] | ||
| + | |||
| + | RefSeq: http://www.ncbi.nlm.nih.gov/refseq/about/ | ||
| + | |||
| + | http://www.yeastgenome.org/ | ||
| + | |||
| + | http://www.ncbi.nlm.nih.gov/refseq/ | ||
| + | |||
| + | Om NCBI Genome Assembly model: http://www.ncbi.nlm.nih.gov/assembly/model/ | ||
| + | |||
| + | http://www.ncbi.nlm.nih.gov/pmc/articles/PMC4702866/ | ||
| + | |||
| + | ==Referansesekvenser== | ||
| + | |||
| + | ===Gjær=== | ||
| + | |||
| + | '''Saccharomyces cerevisiae:''' | ||
| + | |||
| + | S288C: http://www.yeastgenome.org/strain/S288C/overview#resources | ||
| + | |||
| + | http://downloads.yeastgenome.org/sequence/S288C_reference/genome_releases/ | ||
| + | |||
| + | The Reference Genome Sequence of Saccharomyces cerevisiae: Then and Now: http://www.g3journal.org/content/4/3/389.full | ||
| + | |||
| + | |||
| + | '''Brettanomyces (Dekkera) bruxellensis:''' | ||
| + | |||
| + | http://www.ncbi.nlm.nih.gov/genome/11901 | ||
| + | |||
| + | http://www.ncbi.nlm.nih.gov/assembly/GCA_000340765.1 | ||
| + | |||
| + | http://www.ncbi.nlm.nih.gov/pubmed/22663979 | ||
==Søkeverktøy== | ==Søkeverktøy== | ||
| Linje 683: | Linje 1 088: | ||
[http://www.sanger.ac.uk/science/tools/artemis Artemis] | [http://www.sanger.ac.uk/science/tools/artemis Artemis] | ||
| + | |||
| + | ==Andre verktøy== | ||
| + | |||
| + | In silico PCR: https://genome.ucsc.edu/cgi-bin/hgPcr | ||
| + | |||
| + | http://www.complex.iastate.edu/download/Picky/index.html | ||
| + | |||
| + | =Biobanker/artskataloger= | ||
| + | |||
| + | Spanish Type Culture collection (CECT): http://www.cect.org/english/hongos.php | ||
| + | |||
| + | =Organismer= | ||
| + | |||
| + | ==Gjær== | ||
| + | |||
| + | ===Saccharomyces=== | ||
| + | |||
| + | http://www.klikk.no/mat/spise/article1490066.ece | ||
| + | |||
| + | http://wiki.yeastgenome.org/index.php/What_are_yeast%3F | ||
| + | |||
| + | |||
| + | http://www.ncbi.nlm.nih.gov/pmc/articles/PMC3962479/ | ||
| + | |||
| + | The Reference Genome Sequence of Saccharomyces cerevisiae: Then and Now: http://www.straininfo.net/strains/317495 | ||
| + | |||
| + | http://www.ncbi.nlm.nih.gov/genome/?term=txid4932[orgn] | ||
| + | |||
| + | https://www.phys.ksu.edu/gene/chapters.html | ||
| + | |||
| + | http://book.bionumbers.org/what-is-the-macromolecular-composition-of-the-cell/ | ||
| + | |||
| + | 5.8S rRNA: http://yeastmine.yeastgenome.org/yeastmine/report.do?id=1017404&trail=|1017404 | ||
| + | |||
| + | [http://www.yeastgenome.org/browse/?loc=chrXII%3A455382..455603&tracks=DNA%2CAll%20Annotated%20Sequence%20Features%2CDoube_strand_break_hotspots%2CXrn1-sensitive_unstable%20transcripts_XUTs%2CScGlycerolMedia%2C3%27UTRs%2CPolII_occupancy_WT&highlight= 5.8S rRNA (RDN58-2) at SGD] (Lokasjon chrXII:455414..455571) | ||
| + | |||
| + | 5.8S rRNA (RDN58-1) at SgD:http://yeastmine.yeastgenome.org/yeastmine/report.do?id=1017401&trail=|1017401 (Lokasjon: chrXII:455414-455571 reverse strand) | ||
| + | |||
| + | Chromosome XII context is important for rDNA function in yeast: http://nar.oxfordjournals.org/content/34/10/2914.full | ||
| + | |||
| + | ===Brettanomyces=== | ||
| + | |||
| + | NCBI organism page: http://www.ncbi.nlm.nih.gov/genome/?term=txid5007[orgn] | ||
| + | |||
| + | Crauwels et al. Assessing Genetic Diversity among Brettanomyces Yeasts by DNA Fingerprinting and Whole-Genome Sequencing. Appl Environ Microbiol. 2014 Jul; 80(14): 4398–4413. http://www.ncbi.nlm.nih.gov/pmc/articles/PMC4068659/ | ||
| + | |||
| + | Wikipedia: https://en.wikipedia.org/wiki/Brettanomyces_bruxellensis | ||
| + | |||
| + | Partial vinylphenol reductase purification and characterization from Brettanomyces bruxellensis. http://femsle.oxfordjournals.org/content/284/2/213 | ||
| + | |||
| + | https://lup.lub.lu.se/student-papers/search/publication/3632990 | ||
| + | |||
| + | ==Mesoplasma florum== | ||
| + | |||
| + | http://www.ncbi.nlm.nih.gov/genome/?term=Mesoplasma+florum | ||
| + | |||
| + | =Protokoller= | ||
| + | |||
| + | Se https://bitraf.no/wiki/BioHackerLab/Protokoller | ||
| + | |||
| + | [[Category:Biohacking]] | ||
| + | [[Category:Rom]] | ||
Nåværende revisjon fra 23. okt. 2022 kl. 18:32
Biologi gruppen på Bitraf heter Bioraf.
See also our chat channel #biohackers on bitraf.slack.com. To join, send yourself an invitation at https://bitraf.no/slack-invite/
Innhold
- 1 Planlegging
- 2 Avholdte meetups
- 3 Planlagte meetups
- 4 Videre jobbing
- 4.1 Utstyr vi har
- 4.2 Utstyr vi ønsker oss
- 4.3 Mikroskopi
- 4.4 PCR prosjekt
- 4.4.1 Hva er PCR?
- 4.4.2 Hvorfor gjøre PCR?
- 4.4.3 Hva trengs for PCR?
- 4.4.4 Sikkerhetsmomenter:
- 4.4.5 Avfallshåndtering:
- 4.4.6 Reagenser, mulige leverandører og prisoverslag:
- 4.4.7 Artikler/protokoller og lesestoff
- 4.4.8 Possible sub-projects/experiments
- 4.4.9 DIY PCR-maskiner - Eksisterende design og prosjekter
- 4.4.10 Tilgjengelige primere
- 4.4.11 Primers of interest / Shopping list
- 5 Helse, miljø og sikkerheit (HMS)
- 6 Lenker
- 6.1 Lab techniques
- 6.2 Andre grupper og nettsteder
- 6.3 HMS
- 6.4 Lover og forskrifter
- 6.5 Litteratur
- 6.6 Kommersielt tilgjengelig utstyr
- 6.7 Kommersielt tilgjengelige reagenser og forbruksvarer
- 6.8 Leverandører av utstyr, tjenester og forbruksvarer
- 6.9 DIY/Open hardware
- 6.10 Prosjekter til inspirasjon
- 6.11 Diverse
- 7 Bioinformatikk
- 8 Biobanker/artskataloger
- 9 Organismer
- 10 Protokoller
Planlegging
Det har i det siste vært diskusjoner om å starte en egen lab for biologi på Bitraf. Bitraf har allerede medlemmer som jobber med hydroponics/aquaphonics og flere er interessert i ølbrygging. 18 Mai 2016 var det første møtet for folk som er interessert i å starte opp dette. Ønsket er å utvide foreningen med et lokale som egner seg for Gjør-det-selv-biologi og Biologi-hacking som retter seg etter norsk lov og de etiske retningslinjene fremsatt av European DIYbio Congress.
Summary from Bitraf's 1'st MeetUp: https://drive.google.com/file/d/0B5j_-m_-t56rMXNHVVkwVEpUVjg/view
Courses and Equipment: https://docs.google.com/spreadsheets/d/1M2TKaHVBQ3iLuV55A8JCwTCzPXx_aNDFoxyzZBgSM8o/edit#gid=0
Avholdte meetups
- 18. mai 2016: http://www.meetup.com/bitraf/events/230806525/
- 30.mai 2016: http://www.meetup.com/BioHcking/events/231697495/
- 13. juni 2016: http://www.meetup.com/bitraf/events/231692626/ - > Vi fikk besøk av Marius Øgaard fra Oslo Lifetech
- 27. juni 2016: Bruk av DNA-elektroforeseutstyret ble demonstrert, og de fleste fikk prøve pipettering av DNA til gel'en. Heikki presenterte utkast til brev til mulige sponsorer og tok i mot tilbakemeldinger.
- 11. juli 2016: http://www.meetup.com/bitraf/events/232455024/. We performed PCR using 3 different yeast samples. As it became pretty late, the PCR samples were put in the freezer for later electrophoresis and checking of the results. To have something to look at, we performed electrophoresis of samples from the previous two PCR runs. Update: The PCR samples were checked by electrophoresis July 24. 1 of 6 reactions was succesful. See https://bitraf.no/wiki/BioHackerLab/Experimental#11_Jul_2016_-_Bitraf_PCR_.233
- Monday
August 1August 8 2016: DIY Biolab: Using basic bioinformatics Tools; PyMOL: http://www.meetup.com/bitraf/events/232677504/
- Monday August 22: DIY Biolab: A visit from the UiO iGEM team + strawberry DNA extraction: https://www.meetup.com/bitraf/events/233059010/
- Monday September 5 2016: Bioraf byggekveld: http://www.meetup.com/bitraf/events/233816161/ We placed a book shelf in the main working space and put the equipment on it for easier access.
- Monday September 19 2016: Sequence Alignment Workshop: https://www.meetup.com/bitraf/events/234064840/
Planlagte meetups
Videre jobbing
De av deltakerne som ønsket å jobbe med dette videre delte seg i to grupper som skal jobbe med Mikrobiologi og Utstyr til labben. Mikrobiologi-gruppen har planer for å jobbe med ølbrygging og klassifisering av gjær med Open PCR.

Utstyr vi har
- 1x OpenPCR
- Automatpipetter for områdene 1-10, 10-100 og 100-1000 uL. (2 sett)
- 1x lavhastighets mikrosentrifuge for 1.5 mL-rør
- 1x Vortex mixer
- Elektroforese-kammer: 2x Carolina deluxe electrophoresis chamber
- Elektroforesestrømforsyning: 1x BioRad PowerPac Basic.
- 1x DarkReader DR22A blue light transilluminator
- Mikrobølgeovn
- Vanndestillasjonsapparat
- Vannbad
- Varmeplate med magnetrører
- Eppendorf Biophotometer 6131
- Diverse pelistaltiske pumper
- Biohackacademy pelistaltisk pumpe
- NIKON Diaphot TMD mikroskop
Utstyr vi ønsker oss
- pH-meter
- UV-kamera. (Hva brukes dette til? --Jarlemag (diskusjon) 22. jul. 2016 kl. 18:41 (UTC))
- Sentrifuge: Høyhastighetssentrifuge for 10 mL- og 50 mL-rør. Ex. Sorvall Biofuge Primo / Primo R.
- Fryser (ideelt -20 C)
- Kjøleskap
- Varmeskap med shaker. Eks: https://webshop.no.alere.com/222ds-benchtop-shaking-incubator-230v_1.aspx
- Varmeblokk
- Autoklav
- Lab-glass (flasker, rør m.m.)
- Målepipetter i glass
- Flere automatpipetter?
- Filter-utstyr
- Vekst-medier
- Qubit Fluorometer
- Mikroskop med minst 800x forstørrelse (x400 luft, x800 oljeimmersjon), Confokalt og fase kontrast. https://no.wikipedia.org/wiki/Fasekontrastmikroskop f.eks. [1] Optika B-500TiPh fra japanphoto
- Presisjonsvekt (F.eks Kern EMB 100-3: https://www.vektekspert.no/presisjonsvekt-kern-emb.html)
- Mikroplateleser, ex. Tecan Infinite 200
I tillegg trenger vi laboratoriemøbler/inventar som f.eks.:
- Stålbenk/utslagsvask
- Fumehood / labbenk
- Filter til Fumehood
- Oppvaskmaskin
Mikroskopi
Content: Roland van Dierendonck og Arne Andersen (Limnoan)
At Bitraf we have the NIKON Diaphot TMD inverted microscope. Always cover the microscope after using to avoid dust on the lenses. A manual of the NIKON Diaphot TMD can be found here: https://neurophysics.ucsd.edu/Manuals/Nikon/Diaphot-TMD.pdf More links on this webpage from Arizona edu: https://lavinia.as.arizona.edu/~mtuell/scopes/Diaphot.php - according to the author, this instrument was used to create the first test-tube baby!
Fasekontrastmikroskopi: Fasekontrast er en metode for å gjøre mikroskop-preparat bedre synlige uten å farge dem. Rent praktisk brukes sett av matchende ringfilter. Det ene sitter på kondensatoren, det andre inne i objektivet. Filteret på kondensatoren lager en ring av lys. Størrelsen på ringen skal passe med en tilsvarende ring i objektivet. Når alt er riktig innstilt, skal de to ringene overlappe hverandre. Denne innstillingen skal ikke endres av brukeren! Som nevnt, kommer filterne i par: På kondensatoren er de merket med PhL – Ph3. Høyere tall står for høyere forstørrelse på objektivet. Objektivene er merket på tilsvarende måte, men det er vanskelig å se på vårt mikroskop. Vanligvis bytter en filter og objektiv samtidig, men mismatch kan av og til gi interessante effekter. https://en.wikipedia.org/wiki/Phase-contrast_microscopy
Phase-contrast microscopy: Phase-contrast is a method of making microscope specimens more visible without staining them. In practical terms, sets of matching ring filters are used. One is on the condenser, the other inside the lens. The filter on the condenser creates a ring of light. The size of the ring should fit with a corresponding ring in the lens. When everything is set correctly, the two rings should overlap. This setting should not be changed by the user! As mentioned, the filters come in pairs: On the condenser they are marked with PhL - Ph3. Higher numbers represent higher magnification of the lens. The lenses are marked in a similar way, but it is difficult to see on our microscope. Usually, a filter and lens change at the same time, but a mismatch can sometimes give interesting effects. https://en.wikipedia.org/wiki/Phase-contrast_microscopy
Mørkfelt:Mikroskopet på Bitraf har antakelig ikke denne innstillingen. Mørkfelt betyr at en lager en skygge, som dekker hele synsfeltet. På et vanlig mikroskop kan en for eksempel legge en mynt på lampa. Forskningsmikroskop har ofte mørkfelt som et valg sammen med fasekontrast. Når en legger et objekt på mikroskopet, vil noe av lyset brytes, slik at objektene blir lyse på mørk bakgrunn. https://en.wikipedia.org/wiki/Dark-field_microscopy
Dark field: The microscope on Bitraf probably does not have this setting. Dark field means that you create a shadow, which covers the entire field of view. On an ordinary microscope, for example, you can put a coin on the lamp. Research microscopes often have dark fields as a choice along with phase contrast. When you place an object on the microscope, some of the light will be refracted, so that the objects become light on a dark background. https://en.wikipedia.org/wiki/Dark-field_microscopy
Mikroskopkurs
Planlagt av Roland van Dierendonck og Arne Andersen (Limnoan).
En mikroskopkurs varer 2 eller 3 timer og er notvendig for å bruke mikroskop. I kursen vi lære mikroskopdeler, og også å lage mikroskoppreparater.
Kurs inhold:
Hva du IKKE må røre (what NOT to touch! This is important because the microscope is a very expensive and delicate instrument and some parts are better left unchanged):
- Condensator screws
- Condensator focus knob (knotten med ord "TENSION")
- Lenses (don't touch the lenses with your fingers / ikke rør linser)
- Keep the dust cover on when not in use / la støvhette på
Parts to use / Deler vi bruker:
- X&Y-axis knobs / XY-bord bevegelse knot
- diaphragm / blenner
- Phase-contrast condenser / fase-kontrast kondensator
- Fokus knobs (coarse and fine) / fokus knot (grov og fint)
- Binocular eye piece distance / okular
- "Optical path change-over knob" for directing light to ocular or camera
- Front camera port / kameraport
Maintenance / Vedlikehold:
- Both the lenses and the phase-contrast filters are very delicate. Clean by blowing air with a rubber air blower.
Theory Resources (English og Norsk)
- Mikroskopi Presentasjon fra BioHack Academy (PDF): http://biohackacademy.github.io/bha6/class/4/pdf/4.1%20Microscopy%20and%20Optics.pdf
- Enkel Mikroskopi av Arne Andersen (Limnoan) i Biolog (PDF): https://arena-attachments.s3.amazonaws.com/12048616/1b6de336971678994fa83c4be560562e.pdf?1621853707
- En enkel metode for planteplanktonundersøkelse av Arne Andersen (Limnoan): https://arena-attachments.s3.amazonaws.com/12048615/5c491b7bcc512441bb9d35fbaa7e32dd.pdf?1621853700
This is a Nikon microscope and a Nikon camera body can be attached to make photos and films. For other cameras adapters can be designed and 3D printed.
Mikroskoputstyr vi ønsker oss:
- Bedre støvhette
- Rubber air blower
Things to arrange:
- When is the course going to be (date and time)
- Max people that can join (2/3)
- Can we issue a Badge in the P2k16 system of Bitraf so partipants get one when they did the course?
PCR prosjekt
Hva er PCR?
- Polymerase chain reaction/polymerase kjedereaksjon: https://en.wikipedia.org/wiki/Polymerase_chain_reaction
- PCR brukes for å kopiere DNA. En PCR-maskin med reagenser er en "kopi-maskin" for DNA.
- PCR kan brukes til å kopiere opp (amplifisere) DNA fra naturlige kilder/biologisk materiale for videre bearbeidelse eller analyse.
- DNA-fragmenter med en kjent sekvens (rekkefølge på nukleotider, "bokstavene" i DNA) i hver ende velges ut og kopieres selektivt.
- Sammensetning og lengde av DNA-tråden mellom start- og sluttpunktet kan variere. Informajson om lengden av DNA-fragmentene (visualiseres ved gel-elektroforese), og om kopiering fant sted (positiv/negativ reaksjon) kan brukes til å gjøre enkle genetiske analyser.
- Opparbeidet DNA kan (gitt god nok mengde og kvalitet) sendes til nærmere analyse av DNA-sekvensen (sekvensering).
PCR visualization: https://www.youtube.com/watch?v=2KoLnIwoZKU
Hvorfor gjøre PCR?
- En aktivitet med relativt lav terskel, med begrenset behov for opplæring, gode muligheter for å lykkes, overkommelige kostnader og få risikomomenter.
- En klassisk molekylærbiologi-teknikk. I daglig bruk verden over. Uunnværlig for molekylærbiologisk forskning og medisinsk diagnostikk. Nobelpris-vinnende.
- Potensiale for praktiske anvendelser med allmenn interesse. F.eks kontroll av artsopprinnelse for matvarer.
Hva trengs for PCR?
Fast utstyr:
- PCR-maskin/thermocycler. Automatiserer temperaturegulering gjennom reaksjonsforløpet. Temperatur-regulering kan i teorien gjøres manuelt med vannbad ved ulike temperaturer, men dette blir temmelig langsomt og kjedelig
- Mikropipette(r). Fortrinnsvis minst én automatpipette med justerbart volum i området 1-10 μL.
- En eller flere flasker til agarose, ca. 250 mL. Glass eller varmebestandig plast, bør passe i mikrobølgeovn.
- Mikrobølgeovn til oppvarming av agarose, evt. annen varmekilde + magnetrører
- Gel-elektroforesekammer
- Strømforsyning
- Transilluminator m/filter og/eller filterbriller
- Bør ha: Mikrosentrifuge.
- Bør ha: Kjøleskap/fryser til oppbevaring av reagenser (PCR mastermix bør oppbevares frosset) og lage is
- Kjekt å ha: Vanndestillator
- Kjekt å ha: Liten isoporboks eller lignende til å ha is i, for kjøling av prøver under forberedelse.
Forbruksmaterialer:
- Eppendorf-rør (plastrør tilpasset mikrosentrifuge, ca. 1.5 mL)
- PCR-rør (plastrør tilpasset PCR-maskin, ca 0.5 mL)
- Pipette-spisser til automatpipette(r)
Reagenser/kjemikalier:
- PCR mastermix
- TAE/TBE buffer, konsentrert
- DNA-fargestoff (Riktig type med hensyn til transilluminator)
- DNA-ladder (Blanding av DNA-fragmenter med kjent lengde. Brukes som referanse for lengde/størrelse av DNA-fragmenter ved elektroforese.)
- DNA loading dye (Viskøs fargeblanding til utblanding av PCR-produkt før overføring til agarosegel. Kan "hjemmesnekres"?)
- Elektroforese-agarose
- DNA-primere (eksperiment-spesifikke)
- Vann, fortrinnsvis destillert. Evt. flaskevann med lavt mineralinnhold.
Sikkerhetsutstyr:
- Vernebriller
- Engangshansker
- Varmeisolerende hansker e.l. til håndtering av varm agarose
- Fortrinnsvis labfrakk
Sikkerhetsmomenter:
- Strøm gjennom elektroforesekammer (ca. 50-100 V)
- Varm agaroseløsning
- Støtkoking eller glasseksplosjon ved oppvarming av agarose i mikrobølgeovn. Unngås ved å begrense effekt/oppvarmingshastighet, begrense tid under oppvarming og aldri varme opp lukkede flasker/beholdere.
- Potensielt skadelige kjemikalier (eks. Ethidum-bromid, "ETBR") og fargestoff som krever bruk av transilluminator med UV-stråling bør unngås. Alternativer med lavere risiko og bedre miljøprofil bør brukes, f.eks "GelGreen" fargestoff (brukes med transilluminator med synlig blått lys, redusert fare for øye/hudskader).
Avfallshåndtering:
Avhenger av reagensvalg! Individuell vurdering må gjøres for hvert stoff. Generelt:
- Størknet agarose kastes som restavfall. Flytende agarose må ikke tømmes i avløp, da dette vil størkne ved avkjøling.
- Brukte bufferløsninger kan helles i avløp
- Forbruksmateriell av plast med eventuelle reagensrester kastes i restavfall
- Utstyr rengjøres med vann etter bruk
Reagenser, mulige leverandører og prisoverslag:
PCR-reagenser:
DongSheng Biotech: http://dongshengbio.com/en/cpjs.asp?classname=PCR%20Products
Taq Mix: http://dongshengbio.com/en/xxcp.asp?id=330/ http://dongshengbio.com/en/UploadFiles/2012516105050871.pdf
Eksempel-bestilling (2014):
Product Name Cat No Description Qty Unit Unit Price Value
Taq Mix (2x) P2011 1 1ml $8.80 $8.80
Water, nuclease-free P9021 1 5x1ml $2.00 $2.00
PCR and DNA Fragment Purification Kit N1091 1 50preps $20.00 $20.00
50bp ladder M1041 1 50ug $16.00 $16.00
6xDNA Loading Dye M9041 1 5x1ml $4.60 $4.60
shipment viaFedEx $55.00
Total $106.40
DNA-fargestoff:
GelGreen:
https://biotium.com/technology/gelred-gelgreen-nucleic-acid-gel-stains/
GelGreen, Carolina.com: http://www.carolina.com/biotechnology-electrophoresis-reagents/gel-green/217305.pr?question= (ca. $64.50 + shipping / 150 uL 10 000 x konsentrasjon. Nok til ca. 30-40 agarose-geleer.)
Suggested protocols for working with GelGreen: http://embitec.com/downloads/Suggested_Protocols-GelGreen.pdf
GelRed-GelGreen Safety report: http://biotium.com/wp-content/uploads/2013/07/GR-GG-Safety.pdf
Elektroforese-buffer:
TAE (Tris/Acetate/EDTA)-buffer, Promega: https://no.promega.com/products/biochemicals-and-labware/biochemical-buffers-and-reagents/tae-buffer_-molecular-biology-grade-_tris_acetate_edta_/ (390 kr/1000 mL 10x konsentrasjon = 39 kr/L ferdig buffer)
TAE elektroforesebuffer 50 x, Frederiksen Scientific: http://no.frederiksen.eu/shop/product/tae-elektroforesebuffer-50x (1 250 kr/ 500 mL 50x konsentrasjon = 50 kr/L ferdig buffer
TBE (Tris/Borate/EDTA)-buffer kan også brukes.
Elektroforese-agarose:
Agarose, 10 g, Frederiksen Scientific: http://no.frederiksen.eu/shop/product/agarose--10-g (kr 319 + frakt. Nok til ca. 10 agarose-gel'er (Gitt 50 mL 2 % agarose). Temmelig dyrt...Har tidligere kjøpt 100g på eBay for USD 40 + frakt.
DNA-primere: Macrogen Inc:
Ca. 0.2 EUR/basepar (bp) x ca. 30 bp x 2 primere = ca. 12 EUR Shipping ca 20 Sum ca. 30-40 EUR. (2013-priser)
Artikler/protokoller og lesestoff
- The PCR controls you must use: http://bitesizebio.com/4074/the-pcr-controls-you-must-use/
- Yaest colony PCR. Utvalg av protokoller @ OpenWetware: http://openwetware.org/wiki/Yeast_Colony_PCR
- http://www.jove.com/video/3998/polymerase-chain-reaction-basic-protocol-plus-troubleshooting
- http://onlinelibrary.wiley.com/doi/10.1080/15216549700202551/pdf
- Simple And Reliable Procedure For PCR Amplification Of Genomic Dna From Yeast Cells Using Short Sequencing Primers: http://onlinelibrary.wiley.com/doi/10.1080/15216549700202551/pdf
- http://openwetware.org/wiki/Agarose_gel_electrophoresis
- http://no.frederiksen.eu/inspirasjon/biologi/bioteknologi/tips-og-raad
- http://www.naturfag.no/utstyrsbeskrivelse/vis.html?tid=709639
- https://seqcore.brcf.med.umich.edu/sites/default/files/html/pcr.html
DNA Learning Center Biology Animation Library - Polymerase Chain Reaction: https://www.dnalc.org/resources/animations/pcr.html
Possible sub-projects/experiments
In rough order of increasing difficulty/complexity?
- Electrophoresis demonstration/equipment test: Demonstrate/test equipment and reagents for agarose gel electrophoresis. Separate and visualize DNA fragments of known size (DNA ladder).
- PCR demonstration/equipment test: Demonstrate/test equipment and reagents for PCR and agarose gel electrophoresis. Amplify DNA fragment of known expected size from purified DNA or from biological material (yeast?). Visualize and determine size/length of the DNA fragment(s) by agarose gel electrophoresis.
- Animal tissue/foodstuff species identification: Demonstrate/test identification of DNA from a suspected/known species in raw or processed food (ex, horsemeat).
DIY PCR-maskiner - Eksisterende design og prosjekter
https://www.kickstarter.com/projects/563115656/3d-printer-into-pcr-machine-conversion
Arduino PCR thermal cycler for under $85: http://www.instructables.com/id/Arduino-PCR-thermal-cycler-for-under-85/?ALLSTEPS
"Coffee Cup PCR": http://www.instructables.com/id/Coffee-Cup-PCR-Thermocycler-costing-under-350/?ALLSTEPS
http://www.popsci.com/diy/article/2013-04/gene-machine
Tilgjengelige primere
| Navn | Sekvens | Beskrivelse | Lengde | Templat |
|---|---|---|---|---|
| Ec_lld_Rev | GTTTCTTCCTGCAGCGGCCGCTACTAGTAtgcaggtctcctggagtccacgc | REV-primer for E. coli lld promoter + RBS. Se http://2012.igem.org/Team:NTNU_Trondheim/Experiments_and_Results | 52 | E. coli |
| Ec_lld_FWD | GTTTCTTCGAATTCGCGGCCGCTTCTAGAGcacattcctataggccgagtaaggt | FWD-primer for E. coli lld promoter + RBS. | 55 | E. coli |
| Fd2trim | GAGTTTGATCATGGCTCAG | Wide-range bacterial. | ||
| Porcine FWD + Porcine REV | [2] |
MEATF + MEATR [2]
HorseSSR-FWD + HorseSSR-REV [2]:
FWD: 5'-TTC TGC TCT GGG TGT GCT ACT T-3' (22mer) REV: 5'-CTA CTT CAG CCA GAT CAG GC-3' (20mer)
S-D-Bact-0515-a-A-19 + S-D-Bact-0341-b-S-17 [1]
S-D-Bact-0515-a-A-19: 5'-TTA CCG CGG CTG CTG GCA C-3' (19mer) S-D-Bact-0341-b-S-17: 5'-CCT ACG GGN GGC WGC AG-3' (17mer)
[1]: See http://openwetware.org/wiki/User:Jarle_Pahr/16S_RNA
[2]: See http://openwetware.org/wiki/User:Jarle_Pahr/Meat
Primers of interest / Shopping list
Yeasts:
"V9D (5'-TTAAGTCCCTGCCCTTTGTA-3') and LS266 (5'-GCATTCCCAAACAACTCGACTC-3') are used to amplify an 800-1300 bp fragment that encompasses a portion of the 18S and 28S rRNA genes and the entire intervening ITS1, 5.8S and ITS2 rRNA regions." (Todd M Pryce. "Universal Detection and Identification of Fungi by PCR and DNA sequencing" in PCR for Clinical Microbiology, SpringerLink 2010.)
ITS 1 (5' TCCGTAGGTGAACCTGCGG 3') + ITS4 (5' TCCTCCGCTTATTGATATGC 3'). " In the present study, the restriction patterns generated from the region spanning the internal transcribed spacers (ITS1 and ITS2) and the 5.8S rRNA gene were used to identify a total of 132 yeast species belonging to 25 different genera, including teleomorphic and anamorphic ascomycetous and basidiomycetous yeasts." (http://www.ncbi.nlm.nih.gov/pubmed/10028278, full text available through ResearchGate).
ITS1 and ITS4 available (10 USD for 100 reactions) from The ODIN: http://www.the-odin.com/fungal-its-pcr-primers-for-identification-and-barcoding/
E. coli:
rrnB p1_74bp_FWD_R caaccggtgttgcgcggtcagaaaatta rrnB p1_74bp_REV_R gtacatgtagtggtggcgcattatagg
Gives a short fragment.
pSB-M1g/pJP-1 plasmids:
pSB-SeqA/GFP-END-LVA-REV. Sequencing of and/or PCR demonstration using plasmid pSB-M1g. PCR from pSB-M1g without adding LVA tag should give fragment of aprox. 800 bp (size of GFP ORF + ~ 1 bp upstream).
pSB-SeqA: tgcaagaagcggatacag
GFP-END-LVA-REV: agaggatcccttaagttaagctactaaagcgtagttttcgtcgtttgctgctttgtatagttcatccatgcc (LVA sequence can be removed from the 5' end).
For PCR from pJP-1, replace pSB-seqA with pJP-1_seq5 (binding site is upstream of AgeI, as such this site is preserved from pSB-mg1, and this primer combination can also be used with pSB-mg1 for a longer PCR fragment)
Helse, miljø og sikkerheit (HMS)
http://www.ncbi.nlm.nih.gov/books/NBK55884/
El-sikkerhet
https://www.physics.ohio-state.edu/~p616/safety/fatal_current.html
Ved nødsfall
Giftinformasjonsentralen, døgnåpen vakttelefon: 22 59 13 00.
Legevakt: 116 117
Avfallshåndtering
Hva er farlig avfall? Se Forskrift om gjenvinning og behandling av avfall (avfallsforskriften) kapittel 11, Farlig avfall.I henhold til forskriftens §11-2 er farlig avfall definert som
- a) avfall som skal klassifiseres som farlig i henhold til vedlegg 1 til kapitlet,
- b) annet avfall som skal klassifiseres som farlig i henhold til vedlegg 2 nr. 1 til kapitlet.
Avfallsbesitter har ansvaret for å vurdere om avfallet omfattes av bestemmelsene i dette kapitlet.
Vedlegg 1 til forskriftens Kapittel 11 er Den europeiske avfallslisten (EAL). EAL angir at:
Som hovedregel skal avfall fra virksomheter m.m. som nevnt i kapittel 01 til 12 eller 17 til 20 i avfallslisten identifiseres ved hjelp av de sekssifrede avfallskodene i disse kapitlene.
Avfall som skal identifiseres ved hjelp av en avfallskode som er merket med stjerne, skal klassifiseres som farlig avfall med mindre Miljødirektoratet eller den Klima- og miljødepartementet bemyndiger har bestemt noe annet i medhold av § 11-2 tredje ledd.
Vedlegg 2 til forskriftens Kapittel 11angir Kriterier som gjør avfall farlig.
Punkt 1 av Vedlegg 2, Egenskaper som gjør avfall farlig, lyder som følger:
Ved vurderingen av om avfall som nevnt i § 11-2 bokstav b og vedlegg 1 nr. 3 annet ledd bokstav a skal klassifiseres som farlig, gjelder vedlegg III til direktiv 2008/98/EF (engelsk versjon) som endret ved forordning (EU) nr. 1357/2014, med unntak for HP 1, HP 9 og HP 15.(...)
Farlige stoffer:
Begrepet "farlige stoffer" brukes i forskriften. Hva er farlige stoffer? I henhold til forskriftens §11-3 er farlige stoffer definert som
stoffer som skal klassifiseres som farlige i henhold til forordning (EF) nr. 1272/2008 artikkel 3, jf. forskrift 16. juni 2012 nr. 622 om klassifisering, merking og emballering av stoffer og stoffblandinger (CLP).
I henhold til forordning (EF) nr 1272/2008 artikkel 3 er farlige stoffer definert som
A substance or a mixture fulfilling the criteria relating to physical hazards, health hazards or environmental hazards, laid down in Parts 2 to 5 of Annex I is hazardous and shall be classified in relation to the respective hazard classes provided for in that Annex.
Håndtering og levering av farlig avfall:
Det er ikke tillatt å fortynne farlig avfall for å komme under grenseverdiene, jf. DIRECTIVE 2008/98/EC artikkel 7 punkt 4.
Avfallsforskriftens § 11-5 først og andre ledd lyder som følger:
Farlig avfall skal tas hånd om på en forsvarlig måte. Alle som oppbevarer, transporterer eller håndterer farlig avfall, skal treffe nødvendige tiltak for å unngå fare for forurensning eller skade på mennesker eller dyr.
Farlig avfall skal ikke blandes sammen med annet avfall. Ulike typer farlig avfall skal ikke sammenblandes dersom dette kan medføre fare for forurensning, eller skape problemer for den videre håndteringen av avfallet.
Forskriftens §11-8 første ledd lyder som følger:
Virksomhet hvor det oppstår farlig avfall, skal levere dette til den som etter § 11-6 og § 11-7 kan håndtere avfallet, eller til virksomhet utenfor Norge i henhold til reglene om grensekryssende transport av avfall i kapittel 13. Det farlige avfallet skal leveres minst 1 gang pr. år. Plikten inntrer ikke før den totale mengden farlig avfall overstiger 1 kg.
Forskriftens §11-12 første punktum lyder som følger:
Virksomhet som leverer farlig avfall skal gi tilstrekkelige opplysninger om avfallets opprinnelse, innhold og egenskaper, slik at den videre håndteringen av avfallet kan skje på en forsvarlig måte.
Noen avfallskategorier som kan tenkes å være aktuelle:
- 06 02 04 natrium- og kaliumhydroksid
- 06 02 05 andre baser
- 06 01 06 andre syrer
- 20 01 13 løsemidler
- 20 01 14 syrer
- 20 01 15 baser
Aktuelle kjemikalier
Tris
http://echa.europa.eu/substance-information/-/substanceinfo/100.000.969
Faresetninger: H315, H319, H335
Konsentrasjonsgrenseverdier for klassifisering som farlig avfall iht. COMMISSION REGULATION (EU) No 1357/2014 :
H315 & H319: 20%
H335: 20%
Eddiksyre
Faresetninger: H226, H314
Konsentrasjonsgrenseverdier for klassifisering som farlig avfall iht. COMMISSION REGULATION (EU) No 1357/2014 :
H226: N/A. Brannfarlighet må vurderes i hvert enkelt tilfelle.
H314: For farlig avfall klasse HP4 - 1%; For farlig avfall klasse HP8 - 5%
(Når avfall inneholder ett eller flere stoffer klassifisert som Skin corr. 1A, 1B eller 1C (H314) og summen av konsentrasjonene er høyere enn eller lik 5 %, skal avfallet klassifiseres som farlig avfall av typen HP 8.)
Ethylenediaminetetraacetic acid, disodium salt dihydrate (EDTA)
Faresetninger: H332, H373, H302, H315, H319,
Konsentrasjonsgrenseverdier for klassifisering som farlig avfall iht. COMMISSION REGULATION (EU) No 1357/2014 :
H332: 22,5%
H373: 10%
H302: 25%
H315 & H319: 20%
Minste grenseverdi: 10%
DMSO
Guanidinium chloride
Finnes i mange kommersielle kit for DNA-rensing. Typisk konsentrasjon kan være 5 M: http://openwetware.org/wiki/Qiagen_Buffers
http://echa.europa.eu/information-on-chemicals/cl-inventory-database/-/discli/details/52449
Faresetninger (Konsentrasjonsgrenseverdier for klassifisering som farlig avfall iht. COMMISSION REGULATION (EU) No 1357/2014):
- H302: 25%
- H315: 20%
- H319: 20%
Minste grenseverdi: 20%
Basert på dette vil en 5M løsning sannsynligvis regnes som farlig avfall.
Isopropanol
https://echa.europa.eu/information-on-chemicals/cl-inventory-database/-/discli/details/22308
Faresetninger (Konsentrasjonsgrenseverdier for klassifisering som farlig avfall iht. COMMISSION REGULATION (EU) No 1357/2014):
- H225: " If the presence of [the] substance indicates that the waste is flammable, it shall be classified as hazardous by HP 3"
- H319 (20%)
- H336 (no limit given)
Aktuelle stoffblandinger
TAE buffer
Blanding av Tris-Acetate-EDTA. Se aktuelle kjemikalier for grenseverdier for de enkelte stoffene til bruk ved vurdering av klassifisering som farlig avfall. Typiske konsentrasjoner i 50x konsentret buffer kan være:
Tris: ~25% Eddiksyre: ´~6 %. EDTA: <1%.
Jf. begrensende grenseverdier for Tris, Eddiksyre og EDTA basert på disses faresetninger og lik henholdsvis 20%, 1% og 10% vil innholdet av Tris og eddiksyre dermed sannsynligvis medføre at konsentrert, ufortynnet 50X TAE buffer må klassifiseres som farlig avfall ved avhending. Se Aktuelle kjemikalier.
For 10X TAE buffer vil de aktuelle konsentrasjonene være ca.:
Tris: ~5% Eddiksyre: 1,2% EDTA: <0.2%
På grunn av innholdet av eddiksyre vil dermed også 10X TAE buffer sannynligvis klassifiseres som farlig avfall ved avhending.
Konsentrert buffer fortynnes typisk til 1x før bruk. Brukt buffer vil dermed sannsynligvis ikke inneholde konsentrasjoner av de nevnte stoffene over grenseverdiene, og kan i såfall avhendes som vanlig avfall. (OBS: Det er ikke tillatt å fortynne farlig avfall med den hensikt å komme under grenseverdiene).
Påkrevd piktogram iht. CLP: GHS07
Sikkerhetsdatablader
EU-krav til sikkerhetsdatablader er gitt i Annex II til REACH: http://eur-lex.europa.eu/legal-content/en/TXT/PDF/?uri=CELEX:02006R1907-20160401
Se også EHCA Guidance on the compilation of safety data sheets: http://echa.europa.eu/documents/10162/13643/sds_en.pdf
GelGreen: http://biotium.com/wp-content/uploads/2013/07/MSDS-41005.pdf
DSView Nucleic acid stain: https://drive.google.com/open?id=0B9aq85qBYTsWbUJCYTZNTzNHSFFTS1BvdzB0bWZ0NTZjby04
DSBio Taq mix (2x) P2011,P2012: https://drive.google.com/open?id=0B9aq85qBYTsWS1JlUS03V19WUHJGY21OWFVmTHhYSmhIQXVj
DSBio 50 bp ladder: https://drive.google.com/open?id=0B9aq85qBYTsWSDlxR2Vra1ZiUUdFRlpNeFduSHl2TzZnUC1Z
Notis: Inneholder 1-5% 2-amino-2-(hydroxymethyl)-1,3-propanediol, faresetninger Xi, R 36/37/38. (H319/H335/H315) (irriterende). Ufortynnet produkt klassifiseres derfor som farlig avfall klasse HP4., jf. Annex III to Directive 2008/98/EC som revidert ved EU-direktiv 1357/2014
DSBio gel loading dye: https://drive.google.com/open?id=0B9aq85qBYTsWRWk3Qk5jVmJfWlc0NUswTVU4eGVLbk9rS2xB
DSBio NF water: https://drive.google.com/open?id=0B9aq85qBYTsWS1JlUS03V19WUHJGY21OWFVmTHhYSmhIQXVj
National Diagnostics TAE 50x: http://www.nationaldiagnostics.com/msds_pdfs/sds_product10.php?cat_num=EC-872
Merck Millipore agarose for elektroforese: http://www.merckmillipore.com/INTERSHOP/web/WFS/Merck-INTL-Site/en_US/-/USD/ShowDocument-File?ProductSKU=MDA_CHEM-116802&DocumentType=MSD&Language=NO&Country=NO
Faremerking
https://www.pervaco.no/skilt-fundament/ghs-clp-skilt
http://www.erdetfarlig.no/no/Artikler/faremerking/?PageID=74
Konverter fra r-setninger til H-setninger: http://ghs.dhigroup.com/PagesGHS/TranslationTool.aspx
Kjemisk sikkerhet og din virksomhet - informasjonsplakat fra miljødirektoratet: http://www.miljodirektoratet.no/Documents/publikasjoner/M324/M324.pdf
Innføring i klassifisering og merking av kjemikalier: http://www.miljodirektoratet.no/Documents/publikasjoner/M247/M247.pdf
Plakat med oversikt over klassifisering og merking etter CLP: http://www.miljodirektoratet.no/Documents/publikasjoner/M259/M259.pdf
Databaser
http://echa.europa.eu/information-on-chemicals
http://echa.europa.eu/web/guest/information-on-chemicals/cl-inventory-database
Lenker
Lab techniques
Alkaline lysis: http://bitesizebio.com/180/the-basics-how-alkaline-lysis-works/
Andre grupper og nettsteder
http://www.socializedscience.com/projects1.html
BioHack Academy: https://biohackacademy.github.io/
https://www.facebook.com/groups/diybio/
GenSpace (New York): http://genspace.org/
La paillasse (PAris) http://lapaillasse.org/
Biologigaragen (København): http://biologigaragen.org/ & https://www.facebook.com/groups/biologigaragen/
http://www.diybiogroningen.org/
Liste over grupper på DIYbio.org: https://diybio.org/local/
BioHacklabs.org Wiki: http://www.biohacklabs.org/Main_Page
Robert Carlson: http://synthesis.cc
London Biohackspace: http://biohackspace.org/
HMS
WHO Laboratory Biosafety Manual, Third Edition: http://www.who.int/csr/resources/publications/biosafety/Biosafety7.pdf
Biosafety in Microbiological and Biomedical Laboratories 5th Edition, U.S. Department of Health and Human Services: http://www.cdc.gov/biosafety/publications/bmbl5/bmbl.pdf
NTNU - Arbeid med biologiske faktorer: https://innsida.ntnu.no/wiki/-/wiki/Norsk/Arbeid+med+biologiske+faktorer
https://innsida.ntnu.no/wiki/-/wiki/Norsk/Biologiske+faktorer
http://www.reach-chemconsult.com/en/seiten/ghs-konverter.html
http://www.the-scientist.com/?articles.view/articleNo/14477/title/An-Accident-Waiting-to-Happen-/
Elektroforese:
https://web.stanford.edu/dept/EHS/prod/researchlab/lab/safety_sheets/08-136.pdf
http://www.ab.ust.hk/hseo/tips/ls/ls008.htm
http://ehs.unl.edu/sop/s-electrophoresis_safety.pdf
https://www.admin.ox.ac.uk/safety/policy-statements/s11-07/
https://www.admin.ox.ac.uk/safety/policy-statements/s11-07/
http://www.di.uq.edu.au/sparq/RAs/SPARQedDNARNAElectrophoresisRA.pdf
Lover og forskrifter
Lover og forskrifter som er lenket til vil ikke nødvendigvis gjelde for aktiviteter ved Bitraf, men kan likevel brukes som en kilde til HMS-relevant informasjon og veiledning:
Norske forskrifter
Forskrift om særavgifter (relevant mht. bruk av teknisk sprit): http://lovdata.no/forskrift/2001-12-11-1451/§3-3-10
Forskrift om utforming og innretning av arbeidsplasser og arbeidslokaler (arbeidsplassforskriften) - Kapittel 8. Arbeid i omgivelser som kan medføre eksponering for biologiske faktorer: http://lovdata.no/forskrift/2011-12-06-1356/§8-1
Forskrift om tiltaksverdier og grenseverdier for fysiske og kjemiske faktorer i arbeidsmiljøet samt smitterisikogrupper for biologiske faktorer (forskrift om tiltaks- og grenseverdier) : https://lovdata.no/dokument/SF/forskrift/2011-12-06-1358
Forskrift om utførelse av arbeid, bruk av arbeidsutstyr og tilhørende tekniske krav (forskrift om utførelse av arbeid) -Andre del: Krav til arbeid med kjemiske og biologiske risikofaktorer: https://lovdata.no/dokument/SF/forskrift/2011-12-06-1357/KAPITTEL_2#KAPITTEL_2
Forskrift om begrensning i bruk av helse- og miljøfarlige kjemikalier og andre produkter (produktforskriften): https://lovdata.no/dokument/SF/forskrift/2004-06-01-922#KAPITTEL_1
Merk spesielt §5-1:
§ 5-1.Omsetning og import av meget giftige og giftige kjemikalier til privat bruk Til privat bruk er det forbudt å importere kjemikalier merket med faresymbol og farebetegnelse «meget giftig» eller «giftig» i henhold til forskrift om klassifisering, merking mv. av farlige kjemikalier eller som i henhold til forordning (EF) nr. 1272/2008 om klassifisering, merking og emballering av stoffer og stoffblandinger (CLP-forordningen) skal klassifiseres i fareklasse og farekategori Carc. 1A, Carc. 1B, Muta. 1A, Muta. 1B, Repr. 1A, Repr. 1B, Acute Tox. 1, Acute Tox. 2, Acute Tox. 3, STOT RE 1 eller STOT SE 1. Forbudet mot privat import gjelder ikke for motorbensin eller dieselolje til transportformål som innføres på kjøretøyets drivstofftank eller i godkjente reservetanker
https://lovdata.no/dokument/SF/forskrift/2004-06-01-930
REACH & CLP
1272/2008 EC (CLP): http://eur-lex.europa.eu/LexUriServ/LexUriServ.do?uri=OJ:L:2008:353:0001:1355:en:PDF
Forskrift om klassifisering, merking og emballering av stoffer og stoffblandinger (CLP): https://lovdata.no/dokument/SF/forskrift/2012-06-16-622
Forholdet mellom REACH og CLP:
http://echa.europa.eu/addressing-chemicals-of-concern/restrictions/substances-restricted-under-reach
Offisiell tekst: http://eur-lex.europa.eu/legal-content/en/TXT/PDF/?uri=CELEX:02006R1907-20160401
REACH angir plikter både for produsenter/importører og for "downstream users". "Downstream users" er definert som
"companies or individuals:
within the European Union/European Economic Area, who use a substance, either on its own or in a mixture, in their industrial or professional activities."
Antar at våre aktiviteter ikke vil omfattes av denne definisjonen?
Se også http://echa.europa.eu/regulations/reach/downstream-users og http://www.prc.cnrs-gif.fr/reach/en/user_obligations.html
ECHA Navigator: http://echa.europa.eu/support/guidance-on-reach-and-clp-implementation/identify-your-obligations/navigator
http://www.steptoe.com/assets/htmldocuments/Atlee_-_Downstream_Users.pdf
http://echa.europa.eu/documents/10162/966058/tips_users_chemicals_workplace_en.pdf
REACH database: http://echa.europa.eu/information-on-chemicals/registered-substances
Litteratur
Bøker
Biohackers: The Politics of Open Science: http://www.amazon.com/Biohackers-Politics-Science-Alessandro-Delfanti/dp/0745332803/ref=sr_1_3?s=books&ie=UTF8&qid=1463928520&sr=1-3&keywords=biohacker
Open-Source Lab: How to Build Your Own Hardware and Reduce Research Costs: http://www.amazon.com/dp/0124104622/ref=wl_it_dp_o_pC_S_ttl?_encoding=UTF8&colid=2JXTKSS1LI8NT&coliid=I2AIVHRBOC69DXhttp://www.amazon.com/dp/0124104622/ref=wl_it_dp_o_pC_S_ttl?_encoding=UTF8&colid=2JXTKSS1LI8NT&coliid=I2AIVHRBOC69DX
Biopunk: Solving Biotech's Biggest Problems in Kitchens and Garages: http://www.amazon.com/Biopunk-Solving-Biotechs-Problems-Kitchens/dp/1617230073/ref=sr_1_1?s=books&ie=UTF8&qid=1463930140&sr=1-1&keywords=biopunk
The Machinery of Life: http://www.amazon.com/Machinery-Life-David-S-Goodsell/dp/0387849246/ref=pd_sim_14_5?ie=UTF8&dpID=51ZSNcQ3vrL&dpSrc=sims&preST=_AC_UL160_SR106%2C160_&refRID=156THN5QQ1RD2Q4DQ5XB
Illustrated Guide to Home Biology Experiments: http://www.amazon.com/Illustrated-Guide-Home-Biology-Experiments/dp/1449396593?ie=UTF8&psc=1&redirect=true&ref_=oh_aui_detailpage_o08_s00
Techniques in microbiology - a student handbook: http://www.amazon.com/Techniques-Microbiology-Handbook-John-Lammert/dp/0132240114?ie=UTF8&psc=1&redirect=true&ref_=oh_aui_detailpage_o04_s00
Biology Is Technology: The Promise, Peril, and New Business of Engineering Life: http://www.amazon.com/dp/0674060156/ref=rdr_ext_tmb
Exploring Personal Genomics: http://www.amazon.com/Exploring-Personal-Genomics-Joel-Dudley/dp/0199644497?ie=UTF8&psc=1&redirect=true&ref_=oh_aui_detailpage_o06_s00
The Art of Fermentation: An In-Depth Exploration of Essential Concepts and Processes from around the World: http://www.amazon.com/Art-Fermentation-Depth-Exploration-Essential/dp/160358286X?ie=UTF8&psc=1&redirect=true&ref_=oh_aui_detailpage_o06_s00
Budding Yeast: A Laboratory Manual: http://www.cshlpress.com/default.tpl?action=full&--eqskudatarq=1071
Artikler
DIY Bio:
European do-it-yourself (DIY) biology: Beyond the hope, hype and horror: http://www.ncbi.nlm.nih.gov/pmc/articles/PMC4158858/
DIYBIOLOGISTS AS ‘MAKERS’ OF PERSONAL BIOLOGIES: HOW MAKE MAGAZINE AND MAKER FAIRES CONTRIBUTE IN CONSTITUTING BIOLOGY AS A PERSONAL TECHNOLOGY: http://peerproduction.net/issues/issue-2/peer-reviewed-papers/diybiologists-as-makers/?format=pdf
http://blogs.plos.org/synbio/2016/05/03/synbio-democratizing-biotechnology/
Synthetic biology: from mainstream to counterculture.: http://www.ncbi.nlm.nih.gov/pubmed/27316777
DNA barcoding:
http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0066213
Nuclear ribosomal internal transcribed spacer (ITS)region as a universal DNA barcode marker for
Fungi:
Hardware:
Edwin: A Robotic Platform for Automated RNA Extraction and Analysis during Reporter Gene–Based Dynamic Characterization of Bacterial Promoters: http://jla.sagepub.com/content/early/2016/06/17/2211068216655151.long
Nyheter
Kommersielt tilgjengelig utstyr
http://www.edvotek.com/Equipment
Anmeldelser, artikler, etc.: http://www.selectscience.net/
PCR
"MiniPCR" (Samme pris som ferdigbygd OpenPCR, $650. Ikke DIY): http://www.minipcr.com/product-category/equipment-and-accessories/minipcr-thermal-cycler/
OpenPCR: http://openpcr.org/
Elektroforese og transilluminasjon
DarkReader Blue Light Transilluminator: http://www.clarechemical.com/transilluminator.htm
Spektro-/foto-/fluorometri
Qubit 3.0 fluorometer:https://www.thermofisher.com/order/catalog/product/Q33216. Listepris 15 340,00 NOK.
Spektrofotometer V1100D: http://no.frederiksen.eu/nettbutikk/felles-laboratorieutstyr/maaleutstyr/spektrofotometer-v1100d. Listepris 5 995,00 kr eks. MVA.
http://www.ebay.com/itm/Visible-Spectrometer-Laboratory-Spectrophotometer-220V-350-1020nm-721-/401130883021?hash=item5d654387cd:g:ONoAAOSwMmBVoPei. Listepris ca. NOK 2,329.51.
Annet laboratorieutstyr
http://no.frederiksen.eu/shop/product/termostatblokk--labnet--digital--enkel
http://no.frederiksen.eu/shop/product/bakteriedyrkingsskap-cultura
Kommersielt tilgjengelige reagenser og forbruksvarer
Mikrobiologi
Peptone LP0037: http://www.oxoid.com/UK/blue/prod_detail/prod_detail.asp?pr=LP0037
Oxoid yeast extract: http://www.oxoid.com/UK/blue/prod_detail/prod_detail.asp?pr=LP0021&c=UK&lang=EN
PCR
http://www.minipcr.com/product-category/minipcr-learning-labs-and-kits/
Elektroforese og transilluminasjon
https://biotium.com/product/gelgreentm-nucleic-acid-gel-stain-10000x-in-water/
Restriksjonsenzymer
EcoRI:
Gjenkjenningssekvens (plusstråd/komplementær): GAATTC / GAATTC
- http://no.frederiksen.eu/shop/product/restriksjonsenzym-ecori
- https://www.neb.com/products/r0101-ecori
CfoI:
Gjenkjenningssekvens (plusstråd/komplementær): GCGC / GCGC
HaeIII:
Gjenkjenningssekvens (plusstråd/komplementær): GGCC / GGCC
HinfI:
Gjenkjenningssekvens (plusstråd/komplementær): GANTC/GANTC
https://www.neb.com/products/r0155-hinfi
Relevant litteratur:
Clark et al. Extended stability of restriction enzymes at ambient temperatures. Biotechniques. 2000 Sep;29(3):536-8, 540, 542.: http://www.ncbi.nlm.nih.gov/pubmed/10997268
Leverandører av utstyr, tjenester og forbruksvarer
Forbruksvarer og reagenser
http://www.metabion.com/products/index.php
http://dongshengbio.com/en/index.asp
http://www.onlinesciencemall.com/ (Selger bl.a. TAE buffer)
http://www.abpbio.com/product/nucleic-acid-solution-quantitation-kits/
https://www.theconsumablescompany.com/
Oligomersyntese
http://www.sigmaaldrich.com/technical-documents/articles/biology/standard-dna-synthesis.html
Sekvensering
General advice:
http://www.bgi.com/services/genomics/sanger-sequencing/single-sample-sequencing/
http://www.nucleics.com/DNA_sequencing_support/sequencing-service-choosing.html
http://www.nucleics.com/DNA_sequencing_support/sequencing-service-reviews.html
GATC:
LightRun sequencing: https://www.gatc-biotech.com/en/products/sanger-services/lightrun-sequencing.html'
- Prepaid labels
- Premixed DNA and primer
- 4.00 EUR / reaction, minimum order of 100
- (Welcome offer: 50 reactions for 3.50 EUR each)
SUPREMErun sequencing: https://www.gatc-biotech.com/en/products/sanger-services/supremerun-sequencing.html
Macrogen:
http://dna.macrogen.com/eng/support/ces/guide/order_guide.jsp
Standard sequencing: https://dna.macrogen.com/eng/order/ces/std/s_new_step1.jsp
- 5.99 EUR/reaction (express, 24 turnaround)
- 4.5 EUR/reaction (regular, 4-5 workdays turnaround)
- +2 EUR/reaction for purification
EZseq: http://dna.macrogen.com/eng/order/ces/ezseq/ezseq_step1.jsp
- EZseq single direct: 4.5 EUR/label (minimum order of 50).
- 24hr turnaround
Sample purification is not offered with EZseq.
Eco-seq: http://dna.macrogen.com/eng/order/ces/ecoseq/ecoseq_step1.jsp
- Eco-seq single direct: 4.99 EUR/label (minimum order of 50)
- Eco-seq single purification: 6.99 EUR/label
Custom sequencing: https://dna.macrogen.com/eng/support/ces/customized_seq_intro.jsp
Sample preparation guide: https://dna.macrogen.com/eng/support/ces/guide/ces_sample_prep.jsp
From FAQ: "Minimum 20ul of 100ng/ul(plasmids, unpurified PCR products) or 50ng/ul(purified PCR products) are required for a couple of reactions."
Sample submission guide: https://dna.macrogen.com/eng/support/ces/guide/ces_sample_submission.jsp
BaseClear:
See http://www.baseclear.com/genomics/sanger-sequencing
See https://orders.baseclear.com/
Single run sequencing services:
Prepaid barcode sequencing: http://www.baseclear.com/genomics/sanger-sequencing/prepaid-barcode-sequencing
- DNA purification and premixing with primer is done by the customer
Quickshot: http://www.baseclear.com/genomics/sanger-sequencing/quick-shot
Primers options:
- Separate sample and primer or premix possible
- Free usage of universal primers (see standard primer list)
- Custom primer, sent with the order (10 pmol/uL in volume >20 uL (enough for max.10 reactions)
Sample options:
- Bacteria for plasmid isolation (miniprep) on a agar plate or as glycerol stock
- Purified plasmid in a minimal volume of 30 ul with a DNA concentration of 50 – 200 ng/ul
- Purified or Raw PCR product in a minimal volume of 30 ul with a DNA concentration >5 ng/μl
Pricing: ?? Payment: Invoice by e-mail.
Source BioScience:
http://www.lifesciences.sourcebioscience.com/genomic-services/sanger-sequencing-service/
9.5 EUR/reaction for minimum order of 50 (475 EUR)
Concentration requirement for (purified) PCR product: 1ng/µl per 100bp
- Payment by credit card available.
DIY/Open hardware
http://openwetware.org/wiki/DIYbio:Notebook/Open_Gel_Box_2.0
http://hackteria.org/wiki/index.php/DIY_NanoDrop
http://www.thingiverse.com/thing:73910
http://www.instructables.com/id/DIY-BioPrinter/
https://github.com/biohackacademy
Prosjekter til inspirasjon
http://www.instructables.com/id/DIY-Bio-plastics/
Diverse
Bioinformatikk
Genomikk
http://www.ncbi.nlm.nih.gov/projects/genome/assembly/grc/info/definitions.shtml
Databaser
RefSeq: http://www.ncbi.nlm.nih.gov/refseq/about/
http://www.ncbi.nlm.nih.gov/refseq/
Om NCBI Genome Assembly model: http://www.ncbi.nlm.nih.gov/assembly/model/
http://www.ncbi.nlm.nih.gov/pmc/articles/PMC4702866/
Referansesekvenser
Gjær
Saccharomyces cerevisiae:
S288C: http://www.yeastgenome.org/strain/S288C/overview#resources
http://downloads.yeastgenome.org/sequence/S288C_reference/genome_releases/
The Reference Genome Sequence of Saccharomyces cerevisiae: Then and Now: http://www.g3journal.org/content/4/3/389.full
Brettanomyces (Dekkera) bruxellensis:
http://www.ncbi.nlm.nih.gov/genome/11901
http://www.ncbi.nlm.nih.gov/assembly/GCA_000340765.1
http://www.ncbi.nlm.nih.gov/pubmed/22663979
Søkeverktøy
Nedlastbare programmer
Andre verktøy
In silico PCR: https://genome.ucsc.edu/cgi-bin/hgPcr
http://www.complex.iastate.edu/download/Picky/index.html
Biobanker/artskataloger
Spanish Type Culture collection (CECT): http://www.cect.org/english/hongos.php
Organismer
Gjær
Saccharomyces
http://www.klikk.no/mat/spise/article1490066.ece
http://wiki.yeastgenome.org/index.php/What_are_yeast%3F
http://www.ncbi.nlm.nih.gov/pmc/articles/PMC3962479/
The Reference Genome Sequence of Saccharomyces cerevisiae: Then and Now: http://www.straininfo.net/strains/317495
http://www.ncbi.nlm.nih.gov/genome/?term=txid4932[orgn]
https://www.phys.ksu.edu/gene/chapters.html
http://book.bionumbers.org/what-is-the-macromolecular-composition-of-the-cell/
5.8S rRNA: http://yeastmine.yeastgenome.org/yeastmine/report.do?id=1017404&trail=%7C1017404
5.8S rRNA (RDN58-2) at SGD (Lokasjon chrXII:455414..455571)
5.8S rRNA (RDN58-1) at SgD:http://yeastmine.yeastgenome.org/yeastmine/report.do?id=1017401&trail=%7C1017401 (Lokasjon: chrXII:455414-455571 reverse strand)
Chromosome XII context is important for rDNA function in yeast: http://nar.oxfordjournals.org/content/34/10/2914.full
Brettanomyces
NCBI organism page: http://www.ncbi.nlm.nih.gov/genome/?term=txid5007[orgn]
Crauwels et al. Assessing Genetic Diversity among Brettanomyces Yeasts by DNA Fingerprinting and Whole-Genome Sequencing. Appl Environ Microbiol. 2014 Jul; 80(14): 4398–4413. http://www.ncbi.nlm.nih.gov/pmc/articles/PMC4068659/
Wikipedia: https://en.wikipedia.org/wiki/Brettanomyces_bruxellensis
Partial vinylphenol reductase purification and characterization from Brettanomyces bruxellensis. http://femsle.oxfordjournals.org/content/284/2/213
https://lup.lub.lu.se/student-papers/search/publication/3632990
Mesoplasma florum
http://www.ncbi.nlm.nih.gov/genome/?term=Mesoplasma+florum